
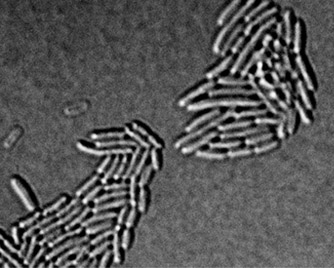
Proteína tóxica secretada por espécies como a Salmonella enterica é usada como arma química contra competidoras, facilitando a colonização do organismo infectado. Estudo pode abrir caminho para o desenvolvimento de novos compostos antimicrobianos (encontro entre duas colônias de bactérias competidoras; imagem: acervo dos pesquisadores)
Proteína tóxica secretada por espécies como a Salmonella enterica é usada como arma química contra competidoras, facilitando a colonização do organismo infectado. Estudo pode abrir caminho para o desenvolvimento de novos compostos antimicrobianos
Proteína tóxica secretada por espécies como a Salmonella enterica é usada como arma química contra competidoras, facilitando a colonização do organismo infectado. Estudo pode abrir caminho para o desenvolvimento de novos compostos antimicrobianos

Proteína tóxica secretada por espécies como a Salmonella enterica é usada como arma química contra competidoras, facilitando a colonização do organismo infectado. Estudo pode abrir caminho para o desenvolvimento de novos compostos antimicrobianos (encontro entre duas colônias de bactérias competidoras; imagem: acervo dos pesquisadores)
André Julião | Agência FAPESP – Pesquisadores da Universidade de São Paulo (USP) apoiados pela FAPESP caracterizaram uma nova família de toxinas antibacterianas presente em bactérias, entre elas a Salmonella enterica. Nessa espécie, a proteína tóxica é usada para matar outras bactérias da microbiota intestinal e facilitar a colonização do intestino de hospedeiros infectados.
O estudo, publicado na Cell Reports, foi destaque na capa da revista.
O membro fundador da nova família é a proteína Tlde1 (sigla para type VI L,D-transpeptidase effector 1), que ataca precursores da parede celular bacteriana. A molécula é secretada por um sistema denominado T6SS (type VI secretion system). Quando a bactéria-alvo é intoxicada, continua crescendo. Mas como sua parede celular está enfraquecida, acaba morrendo devido a um fenômeno chamado pressão osmótica, que faz com que seu conteúdo extravase.
“Essa família de toxinas possui um mecanismo nunca antes descrito. Enquanto outras toxinas antibacterianas secretadas pelo mesmo sistema destroem a parede celular das bactérias-alvo quando ela já está formada, esta atua nos precursores dessa estrutura, prejudicando a sua formação”, explica Ethel Bayer Santos, pesquisadora do Instituto de Ciências Biomédicas (ICB-USP) e coordenadora do projeto apoiado pela FAPESP.
Em um trabalho anterior, a pesquisadora fez parte de uma equipe que descreveu a existência de outro sistema de secreção (T4SS), desta vez em bactérias oportunistas da espécie Stenotrophomonas maltophilia. O artigo também descreveu uma toxina que inibe o crescimento bacteriano, deixando as células-alvo em estado de dormência. O mecanismo de ação da proteína, porém, ainda não foi desvendado (leia mais em: agencia.fapesp.br/32161).
“As bactérias selecionaram essas toxinas durante a evolução e fazem uso delas há milhares de anos, o que as torna potenciais alvos terapêuticos. Podem, futuramente, dar origem a um composto antibacteriano”, diz a pesquisadora.
Mapeamento do genoma
Para encontrar a proteína Tlde1, os pesquisadores analisaram o genoma da Salmonella com foco na região próxima aos genes que codificam as proteínas estruturais do sistema de secreção (T6SS). Encontraram um par de genes com características que indicavam serem codificadores de proteínas com ação antagônica: uma tóxica e outra capaz de neutralizar a toxicidade. No genoma das bactérias, as toxinas antibacterianas geralmente estão localizadas próximas a proteínas que conferem imunidade, para evitar que ocorra autointoxicação.
Para testar a função desse par de genes, os pesquisadores, inicialmente, expressaram aquele que suspeitavam ser responsável pela proteína tóxica em uma bactéria suscetível, a Escherichia coli. A bactéria sobreviveu quando o suposto gene tóxico foi expresso no citoplasma, mas morreu quando a expressão ocorreu no periplasma (estrutura entre o citoplasma e a parede celular), indicando que a toxina tinha como alvo alguma estrutura nesse compartimento da célula bacteriana.
Em seguida, foram expressos no periplasma da E. coli não só a proteína tóxica, mas também a provável proteína de imunidade. A coexpressão neutralizou o efeito tóxico e fez a bactéria sobreviver, confirmando que as duas moléculas são, respectivamente, uma toxina e uma proteína de imunidade.
O sistema de secreção de proteínas T6SS é evolutivamente relacionado ao aparato de bacteriófagos (vírus que infectam bactérias). Ele é composto por 13 proteínas estruturais que se organizam em formato de lança, com uma ponta perfurante envolta por uma bainha contrátil. A célula atacante ejeta a lança cheia de proteínas tóxicas por meio dessa bainha em direção à célula-alvo.
Análises de bioinformática revelaram que membros da família Tlde1 existem em diversas espécies bacterianas, e que essa família provavelmente evoluiu de enzimas bacterianas que participam da síntese da parede celular. O próximo passo da pesquisa é tentar entender, com técnicas de biologia estrutural, como uma enzima que tinha essa função passou a fazer o oposto.
A pesquisa teve como primeiras autoras Stephanie Sibinelli-Sousa e Julia Takuno Hespanhol. Sibinelli-Sousa atualmente realiza mestrado no ICB-USP com bolsa da FAPESP.
Os outros coautores foram Gianlucca Gonçalves Nicastro, Robson Francisco de Souza e Cristiane Rodrigues Guzzo Carvalho, do ICB-USP, e Bruno Yasui Matsuyama, do Instituto de Química (IQ) da USP, além de Stephane Mesnage e Ankur Patel, da Universidade de Sheffield, no Reino Unido.
O artigo A Family of T6SS Antibacterial Effectors Related to l,d-Transpeptidases Targets the Peptidoglycan pode ser lido em: www.cell.com/cell-reports/fulltext/S2211-1247(20)30794-4.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





