
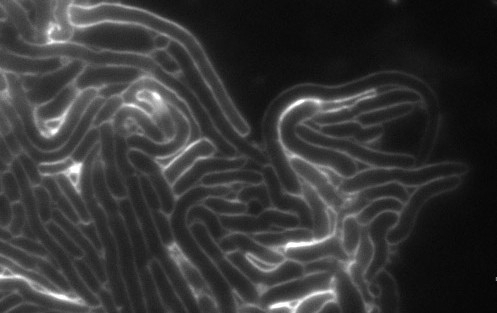
Las proteínas tóxicas perjudican la síntesis de la pared celular de bacterias competidoras. Este hallazgo ayuda a entender de qué manera los patógenos provocan desequilibrios en comunidades microbianas ya establecidas y puede allanar el camino hacia el desarrollo de nuevos compuestos antimicrobianos (imagen de fluorescencia de bacterias intoxicadas/Ethel Bayer-Santos)
Las proteínas tóxicas perjudican la síntesis de la pared celular de bacterias competidoras. Este hallazgo ayuda a entender de qué manera los patógenos provocan desequilibrios en comunidades microbianas ya establecidas y puede allanar el camino hacia el desarrollo de nuevos compuestos antimicrobianos
Las proteínas tóxicas perjudican la síntesis de la pared celular de bacterias competidoras. Este hallazgo ayuda a entender de qué manera los patógenos provocan desequilibrios en comunidades microbianas ya establecidas y puede allanar el camino hacia el desarrollo de nuevos compuestos antimicrobianos

Las proteínas tóxicas perjudican la síntesis de la pared celular de bacterias competidoras. Este hallazgo ayuda a entender de qué manera los patógenos provocan desequilibrios en comunidades microbianas ya establecidas y puede allanar el camino hacia el desarrollo de nuevos compuestos antimicrobianos (imagen de fluorescencia de bacterias intoxicadas/Ethel Bayer-Santos)
Por André Julião | Agência FAPESP – Científicos de la Universidad de São Paulo (USP), en Brasil, caracterizaron a una nueva familia de toxinas antibacterianas presentes en bacterias, entre ellas la Salmonella enterica. En esta especie, la proteína tóxica es utilizada para matar a otras bacterias de la microbiota intestinal y facilitar la colonización del intestino de los hospedantes infectados.
Este estudio, publicado en Cell Reports, apareció destacado en la portada de la revista.
El miembro fundador de la nueva familia es la proteína Tlde1 (las siglas de type VI L, D-transpeptidase effector 1), que ataca a los precursores de la pared celular bacteriana. La misma es secretada a través de un sistema denominado T6SS (type VI secretion system). Cuando la bacteria diana es intoxicada, sigue creciendo, pero como su pared celular se debilita termina muriéndose a causa de un fenómeno denominado presión osmótica, que hace que su contenido se desborde.
“Esta familia de toxinas posee un mecanismo que aún no se había descrito. Mientras que otras toxinas antibacterianas secretadas por el mismo sistema destruyen la pared celular de las bacterias blanco cuando las mismas ya están formadas, esta actúa en los precursores, perjudicando su formación”, explica Ethel Bayer-Santos, investigadora del Instituto de Ciencias Biomédicas (ICB) de la USP y coordinadora del estudio, que cuenta con el apoyo de la FAPESP.
En un trabajo anterior, la científica integró un equipo que describió la existencia de otro sistema de secreción (T4SS), en ese caso en bacterias oportunistas de la especie Stenotrophomonas maltophilia. En el artículo también se describió una toxina antibacteriana que inhibe el crecimiento bacteriano y deja a las células blanco en un estado de latencia. Pero el mecanismo de acción de la proteína aún no ha sido develado (lea más en: agencia.fapesp.br/32407).
“Dado que las bacterias han venido seleccionando a esas toxinas durante la evolución y hacen uso de ellas desde hace miles de años, esto revela potenciales blancos terapéuticos. Las toxinas bacterianas siempre pueden poseer potencial biotecnológico y convertirse en compuestos antibacterianos en el futuro”, dice la investigadora.
La búsqueda
Para hallar la proteína Tlde1, los investigadores analizaron el genoma de la Salmonella en la zona cercana a los genes que codifican proteínas estructurales del sistema de secreción (T6SS). Y encontraron un par de genes con características que indicaban que se trataba de una proteína tóxica y otra que dotaba de inmunidad. En el genoma de las bacterias, las toxinas antibacterianas generalmente se ubican cerca de proteínas que confieren inmunidad, a los efectos de evitar la autointoxicación de la bacteria que la produce.
Para poner a prueba la función de ese dúo de genes, los científicos primeramente expresaron el gen que sospechaban que era responsable de la proteína tóxica en una bacteria susceptible, la Escherichia coli. La bacteria sobrevivió cuando el probable gen tóxico quedó expresado en el citoplasma, pero murió cuando la expresión ocurrió en el periplasma, lo cual indicó que la toxina tenía como blanco alguna estructura de ese compartimento de la célula bacteriana.
Luego se expresaron en el periplasma de la E. coli no solamente la proteína tóxica sino también la probable proteína de inmunidad. La coexpresión neutralizó el efecto tóxico e hizo que la bacteria sobreviviera, confirmando así que ambas proteínas son una toxina y una proteína de inmunidad respectivamente.
El sistema de secreción de proteínas T6SS está relacionado evolutivamente con el aparato de bacteriófagos (virus que infectan a bacterias). Está compuesto por 13 proteínas estructurales que se organizan en formato de lanza, con una punta perforante envuelta en una vaina contráctil. La célula atacante eyecta la lanza llena de proteínas tóxicas con la referida vaina en dirección hacia la célula blanco.
Análisis bioinformáticos revelaron que los miembros de la familia Tlde1 existen en diversas especies bacterianas, y que esta familia probablemente evolucionó a partir de enzimas bacterianas que participan en la síntesis de la pared celular. El próximo paso de la investigación, utilizando biología estructural, consistirá en intentar entender de qué manera una enzima que tenía esa función pasó a hacer lo contrario.
Las primeras autoras de la investigación son Stephanie Sibinelli-Sousa y Julia Takuno Hespanhol, que contaron con sendas becas de iniciación a la investigación científica (expedientes 18/13819-1 y 18/25316-4, respectivamente) de la FAPESP. Sibinelli-Sousa actualmente cursa una maestría en el ICB-USP, también con beca de la FAPESP.
Los otros coautores fueron Gianlucca Gonçalves Nicastro, Robson Francisco de Souza y Cristiane Rodrigues Guzzo Carvalho, del ICB-USP, y Bruno Yasui Matsuyama del Instituto de Química (IQ) de la USP, aparte de Stephane Mesnage y Ankur Patel, de la Universidad de Sheffield, en el Reino Unido.
Puede leerse el artículo intitulado A Family of T6SS Antibacterial Effectors Related to l,d-Transpeptidases Targets the Peptidoglycan en el siguiente enlace: www.cell.com/cell-reports/fulltext/S2211-1247(20)30794-4.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





