

Entender como microrganismos regulam o controle e a produção dessas enzimas é considerado fundamental para a produção de etanol de segunda geração
Entender como microrganismos regulam o controle e a produção dessas enzimas é considerado fundamental para a produção de etanol de segunda geração
Entender como microrganismos regulam o controle e a produção dessas enzimas é considerado fundamental para a produção de etanol de segunda geração
Entender como microrganismos regulam o controle e a produção dessas enzimas é considerado fundamental para a produção de etanol de segunda geração
Elton Alisson | Agência FAPESP – Para produzir o etanol de segunda geração (2G) é preciso realizar a hidrólise enzimática, processo no qual enzimas produzidas por microrganismos atuam em conjunto para degradar e converter carboidratos da palha e do bagaço da cana-de-açúcar em açúcares capazes de sofrer fermentação.
O entendimento dos mecanismos genéticos que regulam o controle e a produção dessas enzimas hidrolíticas por esses microrganismos é considerado fundamental para melhorar as tecnologias voltadas para essa finalidade.
Um grupo de pesquisadores da Universidade Estadual de Campinas (Unicamp), em parceria com colegas do Laboratório Nacional de Ciência e Tecnologia do Bioetanol (CTBE) do Centro Nacional de Pesquisa em Engenharia e Materiais (CNPEM) e da Universidade do Estado do Rio de Janeiro (UERJ), deu um importante passo para compreender diferentes mecanismos biológicos por trás do controle e produção de enzimas hidrolíticas especificamente por fungos.
Realizado no âmbito de um projeto apoiado pela FAPESP, o estudo foi publicado na revista Scientific Reports.
“Nossas descobertas podem ser úteis para o desenvolvimento de enzimas com utilização em coquetéis enzimáticos para a produção de etanol de segunda geração e de outros produtos”, disse Anete Pereira de Souza, professora da Unicamp e coordenadora do projeto, à Agência FAPESP.
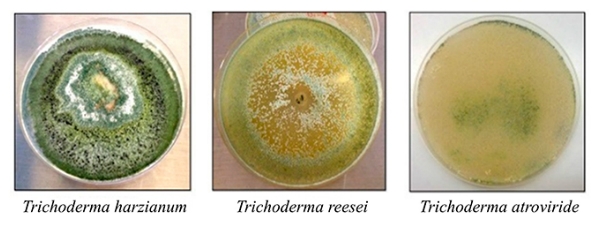
Os pesquisadores analisaram os mecanismos genéticos envolvidos na secreção e expressão de enzimas usadas por fungos das espécies Trichoderma harzianum, Trichoderma ressei e Trichoderma atroviride para degradar a cana.
Encontrados no solo, esses fungos, que crescem em madeiras, cascas e até mesmo em outros fungos e substratos, hidrolisam diferentes tipos de carboidratos – como a celulose do bagaço e da palha da cana – por meio de enzimas presentes em suas paredes celulares.
A fim de avaliar se as enzimas produzidas pelas três espécies de fungos pertencentes ao gênero Trichoderma possuem semelhanças ou diferenças que podem aumentar ou diminuir sua eficiência na degradação de biomassa e se atuam de forma sinérgica durante esse processo, os pesquisadores utilizaram diferentes abordagens de biotecnologia e bioinformática.
Primeiramente, avaliaram o nível de atividade das enzimas secretadas pelas três espécies de fungos durante a fermentação de extratos de bagaço, celulose pura e glicose da cana, por meio da quantidade de proteínas presentes nesses três diferentes substratos no momento de pico do processo de biodegradação.
Por meio de um técnica de biotecnologia chamada RNA-seq, foi possível determinar também os genes expressos. Com base em ferramentas de bioinformática, os pesquisadores compararam esses dados e conseguiram identificar redes de genes corregulados pelas três espécies de fungos que podem ser fundamentais para a biodegradação da biomassa por esses microrganismos.
“Conseguimos identificar redes de corregulação de genes, com alto grau de sinergia, que estão envolvidas na realização do processo de degradação da biomassa da cana pelas três espécies de fungos que codificam enzimas”, disse Jaire Alves Ferreira Filho, doutorando em genética e biologia molecular na Unicamp e um dos autores do estudo.
Alto grau de sinergia
Foram identificados um conjunto de 80 proteínas e seus respectivos genes compartilhados pelas três espécies de fungos. Desse total, 19 proteínas foram observadas em todas as três espécies de fungos.
Essas 19 proteínas e seus respectivos genes detectados estão envolvidos na produção e secreção de enzimas hidrolíticas e estão relacionados a diferentes mecanismos biológicos de biodegradação da biomassa pelas três espécies diferentes de fungos, explicam os pesquisadores.
A elucidação das relações genéticas entre esses conjuntos de genes pode fornecer informações importantes para o desenvolvimento de microrganismos de interesse industrial e também contribuir para a compreensão das reações sinérgicas entre enzimas, destacam os autores do estudo.
“A determinação dessas reações deve promover avanços consideráveis e fornecer uma base sólida para utilização da informação genética para a produção de biocombustíveis e inúmeros biocompostos”, disse Maria Augusta Crivelente Horta, primeira autora do estudo, que realizou pós-doutorado com Bolsa da FAPESP.
O artigo Network of proteins, enzymes and genes linked to biomass degradation shared by Trichoderma species (doi: 10.1038/s41598-018-19671-w), de Maria Augusta Crivelente Horta, Jaire Alves Ferreira Filho, Natália Faraj Murad, Eidy de Oliveira Santos, Clelton Aparecido dos Santos, Juliano Sales Mendes, Marcelo Mendes Brandão, Sindelia Freitas Azzoni e Anete Pereira de Souza, pode ser lido na revista Scientific Reports em www.nature.com/articles/s41598-018-19671-w.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





