

Es un sistema desarrollado por investigadores brasileños que indica en menos de un minuto la presencia del virus en muestras de secreciones nasofaríngeas extraídas con cotonetes.
Es un sistema desarrollado por investigadores brasileños que indica en menos de un minuto la presencia del virus en muestras de secreciones nasofaríngeas extraídas con cotonetes.
Es un sistema desarrollado por investigadores brasileños que indica en menos de un minuto la presencia del virus en muestras de secreciones nasofaríngeas extraídas con cotonetes.
Es un sistema desarrollado por investigadores brasileños que indica en menos de un minuto la presencia del virus en muestras de secreciones nasofaríngeas extraídas con cotonetes.
Por Elton Alisson | Agência FAPESP – Investigadores de la Universidade São Francisco (USF), con sede en la localidad de Bragança Paulista (en el estado de São Paulo, Brasil), desarrollaron una tecnología que permite detectar en menos de un minuto el SARS-CoV-2 directamente en los hisopos nasales que se emplean para tomar muestras de secreciones nasofaríngeas destinadas a la realización de pruebas de diagnóstico del COVID-19.
El desarrollo de este sistema, que tuvo el apoyo de la FAPESP, contó con la colaboración de colegas de la Universidade Presbiteriana Mackenzie (también en Brasil) y de la Universidad de Texas en Austin, Estados Unidos. Este proyecto aparece descrito en un artículo publicado en la revista Analytical Chemistry.
“Este nuevo método permite efectuar el análisis directamente en los hisopos y la obtención del resultado en 45 segundos. De este modo, hace posible la clasificación rápida de pacientes con COVID-19”, dice Andréia de Melo Porcari, docente de la USF y una de las coordinadoras del proyecto.
La captación de moléculas biológicas
Esta tecnología surge como derivación de un sistema de detección y diagnóstico del cáncer desarrollado por la investigadora brasileña Lívia Eberlin en la Universidad de Texas en Austin, basado en espectrometría de masas, una técnica que permite distinguir sustancias en muestras biológicas de acuerdo con su masa molecular.
En dicho método, denominado MasSpec Pen, se utiliza un dispositivo elaborado en plástico con forma de bolígrafo y esterilizable para recolectar moléculas biológicas en la superficie de una muestra de tejido.
La “tinta” del bolígrafo está compuesta de agua, utilizada como solvente para extraer moléculas de una superficie de muestra de tejido, que se transportan a un espectrómetro de masas para su análisis. Con base en algoritmos de aprendizaje de máquinas y modelos estadísticos, el sistema es capaz de indicar si la muestra de tejido analizada contiene células cancerosas.
“Basta con que el bolígrafo toque el tejido para que el agua presente en la punta del dispositivo extraiga las moléculas que se analizarán”, afirma Eberlin.
Los resultados de los estudios clínicos iniciales indicaron que el sistema logró distinguir varios tejidos cancerígenos, incluidos tejidos tumorales de tiroides, mama, pulmón y ovario con una precisión general del 96,3 % con respecto a sus equivalentes normales.
“La idea es que el sistema ayude a los patólogos y a los cirujanos a detectar más rápidamente tejidos cancerígenos y a tomar decisiones más precisas de tratamiento”, dice Eberlin.
La detección del SARS-CoV-2
Con el surgimiento de la pandemia de COVID-19, los investigadores tuvieron a idea de adaptar esta tecnología para detectar el SARS-CoV-2 directamente en los frotis nasofaríngeos extraídos con hisopos. Para ello se hizo necesario realizar adaptaciones en el diseño y en los solventes de la birome.
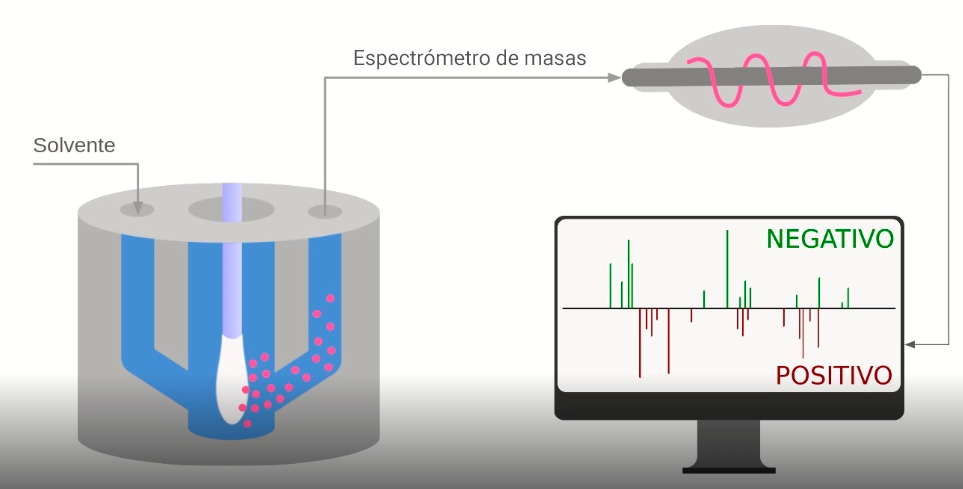
Como el dispositivo solamente toca una pequeña superficie de una muestra de tejido y el material extraído con hisopos queda disperso, los investigadores decidieron invertir el bolígrafo para que el cotonete nasal pudiese introducirse enteramente en una cámara, como la caña de un bolígrafo.
En el interior de la cámara, el hisopo entra en contacto con una pequeña concentración de cloroformo-metanol, utilizado como solvente para extraer las moléculas de las secreciones nasofaríngeas. Las moléculas son succionadas por un orificio existente en la cámara hacia un espectrómetro de masas para su análisis y la detección de la presencia de lípidos que sirven como marcadores que indican se hay o no virus en la muestra.
“Todo este proceso dura menos de un minuto, con el análisis inclusive. Es un ciclo muy rápido, con etapas operativas mínimas y sin necesidad de emplear ningún reactivo especializado”, sostiene Porcari.
La validación de la tecnología
Para validar el método, inicialmente se analizaron hisopos nasofaríngeos de 244 pacientes atendidos en el Hospital Bragantino y en el Complejo Hospitalario Santa Casa Bragança Paulista al comienzo de la pandemia de COVID-19.
Con base en los análisis, fue posible identificar los perfiles lipídicos de esos pacientes y generar clasificadores estadísticos para diferenciar entre individuos sintomáticos positivos, sintomáticos negativos y asintomáticos negativos.
Los resultados del estudio indicaron que los perfiles lipídicos detectados directamente en frotis nasofaríngeos empleando el nuevo método permiten efectuar el triaje rápido de pacientes con COVID-19.
“Este nuevo método de análisis de hisopos puede adaptarse para la detección de muchas otras infecciones víricas y bacterianas y para realización de otros exámenes, como la prueba de Papanicolaou [para la prevención del cáncer de cuello uterino]”, afirma Eberlin.
Los investigadores pretenden realizar ahora una validación interlaboratorios en colaboración con la Universidade Presbiteriana Mackenzie.
“La idea es demostrar con muestras independientes que este nuevo método de análisis es válido, independientemente de los laboratorios, los equipos y los analistas que realicen el test”, explica Porcari.
El artículo Rapid screening of COVID-19 directly from clinical nasopharyngeal swabs using the MasSpec Pen (DOI: 10.1021/acs.analchem.1c01937), de Kyana Y. Garza, Álex A. Rosini Silva, Jonas R. Rosa, Michael F. Keating, Sydney C. Povilaitis, Meredith Spradlin, Pedro H. Godoy Sanches, Alexandre Varão Moura, Junier Marrero Gutierrez, John Q. Lin, Jialing Zhang, Rachel J. DeHoog, Alena Bensussan, Sunil Badal, Danilo Cardoso de Oliveira, Pedro Henrique Dias Garcia, Lisamara Dias de Oliveira Negrini, Marcia A. Antonio, Thiago C. Canevari, Marcos N. Eberlin, Robert Tibshirani, Livia S. Eberlin y Andreia M. Porcari, puede ser leído en la revista Analytical Chemistry, en el siguiente enlace: https://pubs.acs.org/doi/10.1021/acs.analchem.1c01937?ref=pdf&.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





