

Científicos de un centro de investigación que cuenta con el apoyo de la FAPESP, en Brasil, estudiaron cómo se forma la principal enzima implicada en la multiplicación del nuevo coronavirus dentro de las células. Y sus hallazgos se describen en el Journal of Molecular Biology (cristal de la proteína 3CL del SARS-CoV-2; foto: CNPEM)
Científicos de un centro de investigación que cuenta con el apoyo de la FAPESP, en Brasil, estudiaron cómo se forma la principal enzima implicada en la multiplicación del nuevo coronavirus dentro de las células. Y sus hallazgos se describen en el Journal of Molecular Biology
Científicos de un centro de investigación que cuenta con el apoyo de la FAPESP, en Brasil, estudiaron cómo se forma la principal enzima implicada en la multiplicación del nuevo coronavirus dentro de las células. Y sus hallazgos se describen en el Journal of Molecular Biology
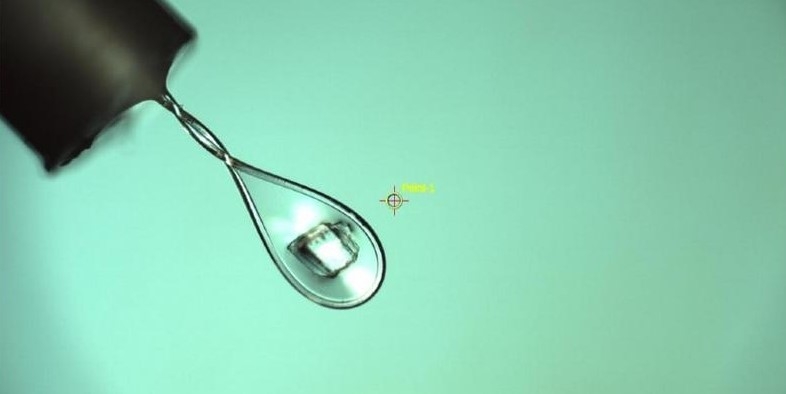
Científicos de un centro de investigación que cuenta con el apoyo de la FAPESP, en Brasil, estudiaron cómo se forma la principal enzima implicada en la multiplicación del nuevo coronavirus dentro de las células. Y sus hallazgos se describen en el Journal of Molecular Biology (cristal de la proteína 3CL del SARS-CoV-2; foto: CNPEM)
Por Maria Fernanda Ziegler | Agência FAPESP – Científicos ligados al Centro de Investigaciones e Innovación en Biodiversidad y Fármacos (CIBFar), en Brasil, develaron detalles del proceso de maduración de la principal enzima implicada en la replicación del nuevo coronavirus, conocida como 3CL. Este descubrimiento, que aparece descrito en el Journal of Molecular Biology, facilita la búsqueda de medicamentos que puedan impedir de entrada este proceso.
“Al cabo de un año y medio de pandemia, de mínima contamos con media docena de vacunas con aplicación clínica, pero seguimos careciendo de fármacos con probada eficacia y seguridad. El desarrollo de antivirales es efectivamente más difícil. Así y todo, aun cuando tenemos buenos inmunógenos, la obtención de un medicamento para el COVID-19 sigue siendo sumamente importante, en caso de que el virus le escape a las vacunas”, afirma Glaucius Oliva, coordinador del CIBFar, un Centro de Investigación, Innovación y Difusión (CEPID) de la FAPESP con sede en el Instituto de Física de São Carlos, de la Universidad de São Paulo (IFSC-USP).
En el referido artículo, los científicos describen el mecanismo molecular mediante el cual la principal proteasa del SARS-CoV-2 –la enzima responsable de la multiplicación del virus– se autoprocesa y se vuelve activa para replicar el material genético del patógeno (el ARN) dentro de las células hospedantes.
“Cuanto más entendemos el metabolismo del virus y sus etapas de replicación, más fácilmente logramos vislumbrar blancos en este proceso, con miras a poder desarrollar moléculas capaces de frenarlo de entrada”, dice Gabriela Noske, doctoranda del CIBFar y primera autora del artículo.
Según Oliva, se trata de un estudio de ciencia básica, pero con aplicaciones inmediatas. “A diferencia de lo que observamos en otros virus, tales como el del Zika, el del dengue o el de la fiebre amarilla, en el nuevo coronavirus la proteasa no actúa en forma monomérica [como una molécula aislada]. Para que la misma se active y pase a multiplicar el ARN del SARS-CoV-2, debe ser dimérica, es decir que es necesario que haya un par de copias de la proteasa para que esta pueda cortarse a sí misma y a las otras proteínas responsables del metabolismo del virus dentro de las células”, explica.
Oliva se encuentra a la cabeza de un proyecto multidisciplinario que cuenta con el apoyo de la FAPESP, y que congrega a investigadores del Instituto de Ciencias Biomédicas (ICB-USP), del Instituto de Química de São Carlos (IQSC-USP), de la Facultad de Ciencias Farmacéuticas de Ribeirão Preto (FCFRP-USP), de la Universidade Estadual Paulista (Unesp) y de la Universidad de Campinas (Unicamp), abocado a la búsqueda de antivirales para el tratamiento del COVID-19 (lea más en: agencia.fapesp.br/33536/)
Son múltiples etapas
El ARN del SARS-CoV-2 está protegido por una “envoltura” formada por lípidos y proteínas, entre ellas la famosa spike o espícula, que compone la estructura en forma de corona que le da su nombre a la familia Coronaviridae. Cuando el virus invade las células y es engolfado por estas, se libera el ARN de la referida cápsula.
Dentro de las células, el principal objetivo del virus es multiplicarse. En esta fase, las proteínas estructurales dejan de ejercer su función fundamental (el transporte del ARN y la evasión del sistema inmunitario) y entran en escena las llamadas proteínas no estructurales, responsables del metabolismo vírico dentro del hospedante.
“El microorganismo necesita efectuar copias de su ARN. Como no cuenta con todos los mecanismos necesarios para ese fin, debe secuestrar algunas funciones de las células que invade. Otras funciones metabólicas, específicas del virus, les competen a las proteínas no estructurales, tales como la proteasa principal y otras 15 moléculas. Nuestro estudio se enfocó en la proteasa principal”, comenta André Godoy, coautor del artículo e investigador del IFSC-USP.
Godoy explica que, mientras que las proteínas estructurales suelen servir como blanco para el desarrollo de vacunas, las no estructurales se utilizan como referencia para la elaboración de medicamentos antivirales. Este es el caso de los cócteles utilizados en el tratamiento del sida, que tienen en la proteasa del VIH a uno de sus blancos.
El descubrimiento de que la proteasa principal del nuevo coronavirus pasa por diferentes fases hasta alcanzar su madurez, para entonces favorecer la multiplicación del SARS-CoV-2 en las células infectadas, solamente fue posible merced a una investigación realizada en la más compleja estructura científica de Brasil.
El año pasado, el experimento a cargo de Godoy y de la investigadora Aline Nakamura inauguró la primera estación de investigación de Sirius, el acelerador de partículas de última generación cuya puesta a punto está ultimándose en el Centro Nacional de Investigaciones en Energía y Materiales (CNPEM), con sede en la ciudad paulista de Campinas (lea más en: agencia.fapesp.br/34533/).
En tres días, y con la ayuda de un potente haz de luz sincrotrón, fue posible determinar la estructura de más de 200 cristales de dos proteínas del nuevo coronavirus. Entre ellas se encontraba la proteasa principal, en diversas formas y en complejos con varios ligandos.
“Junto con la spike, la proteasa principal es la proteína más estudiada del nuevo coronavirus. Hasta ahora no se sabía de qué manera procesa el SARS-CoV-2 dos copias de esta enzima para crear en su estructura el área a la que se conoce con el nombre de ‘sitio activo’, donde logra procesar a las otras proteínas sintetizadas con base en la información contenida en el genoma viral. Existen otros tipos de virus que también poseen esta característica, pero la innovación en este estudio radica en haber comprendido cómo sucede esto”, informa Godoy.
Los investigadores explican que, al ser un virus de ARN, el SARS-CoV-2 ya llega a las células listo para su transcripción a cargo de los orgánulos llamados ribosomas, para entonces producir proteínas no estructurales.
Con todo, como el SARS-CoV-2 posee una sola cadena de ARN, antes es necesario decodificar todas las proteínas no estructurales (y estructurales) en incontables oportunidades. Para ello, el virus las produce como una sola y larga proteína (poliproteína), que luego debe romperse para formar las 16 moléculas encargadas de los mecanismos metabólicos.
“Al ser una estructura mucho más sencilla, el ARN del virus codifica a todas las proteínas juntas, pegaditas, como si conformasen un largo collar de cuentas. Las elabora en los ribosomas de las células invadidas bajo la forma de una larga poliproteína que deberá cortar en trozos. Pero existe un problema: la que corta las proteínas es la proteasa principal, que también se encuentra presente en ese ‘collar de cuentas’. Por ende, esta debe hallar la forma de cortarse a sí misma y, con base en esta acción, escindir a las otras”, explica Oliva.
Los análisis con el haz de luz sincrotrón –un tipo de radiación electromagnética sumamente brillante y muy empleada en estudios de biología estructural– les permitieron a los investigadores detectar de qué manera lleva a cabo este proceso la proteasa principal. Este trabajo reveló que, al fragmentar sus dos puntas, dicha proteasa principal modifica su estructura.
“Asimismo, demostramos que, para el procesamiento de una de las puntas [denominada C-terminal], la enzima necesita contar con un socio dímero, es decir, con una proteína igual que logre fragmentar la parte delantera. Esta logra fragmentar una punta sola, pero no la otra. En tanto, el socio [la otra proteína madura] se unirá a esa parte para fragmentar luego lo que falta”, informa Godoy.
El proceso de maduración de la proteasa principal, según explica el investigador, permite que esta salga de un estadio en el que forma parte de una larga cadena de proteínas, logre autofragmentarse en la parte N-terminal, encontrarse con otra cadena dentro de la célula, formar un dímero y efectuar el procesamiento de la parte C-terminal, formando así por último su estructura madura y activa.
Una luz al final del túnel
De acuerdo con el coordinador del CIBFar, la farmacéutica Pfizer está realizando ensayos clínicos con un medicamento que puede bloquear a la proteasa principal, pero en su fase madura. La farmacéutica Merck también cuenta con un estudio clínico con una molécula que bloquea a otra proteína no estructural llamada polimerasa, responsable de sintetizar copias del ARN viral.
Oliva hace hincapié en que cualquier fármaco antiviral actúa encastrándose en un receptor. “El mundo entero se encuentra abocado a la búsqueda de drogas candidatas que se encajen en la proteasa, pero mirando hacia la estructura de la enzima ya lista, madura. Lo que demostramos es que existen variaciones de estadios anteriores de esa proteasa que pueden erigirse como blancos más interesantes para el desarrollo de fármacos. Es como cortar la maleza antes de que crezca”, concluye.
Puede leerse el artículo intitulado A Crystallographic Snapshot of SARS-CoV-2 Main Protease Maturation Process en el siguiente enlace: www.sciencedirect.com/science/article/pii/S0022283621003429.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





