

Sistema desenvolvido por pesquisadores brasileiros indica em menos de um minuto a presença do vírus em amostras de secreções nasofaríngeas coletadas por meio de swabs
Sistema desenvolvido por pesquisadores brasileiros indica em menos de um minuto a presença do vírus em amostras de secreções nasofaríngeas coletadas por meio de swabs
Sistema desenvolvido por pesquisadores brasileiros indica em menos de um minuto a presença do vírus em amostras de secreções nasofaríngeas coletadas por meio de swabs
Sistema desenvolvido por pesquisadores brasileiros indica em menos de um minuto a presença do vírus em amostras de secreções nasofaríngeas coletadas por meio de swabs
Elton Alisson | Agência FAPESP – Pesquisadores da Universidade São Francisco (USF), em colaboração com colegas da Universidade do Texas em Austin, nos Estados Unidos, e da Universidade Presbiteriana Mackenzie, desenvolveram uma tecnologia que permite detectar em menos de um minuto o SARS-CoV-2 diretamente de swabs (cotonetes) nasais, empregados para coletar amostras de secreções nasofaríngeas para a realização de teste para diagnóstico de COVID-19.
O sistema, desenvolvido por meio de projeto apoiado pela FAPESP, foi descrito em um artigo publicado na revista Analytical Chemistry.
“O novo método permite a análise direta de swabs e a obtenção do resultado em 45 segundos. Dessa forma, possibilita a triagem rápida de pacientes com COVID-19”, diz Andréia de Melo Porcari, professora da USF e uma das coordenadoras do projeto.
Coleta de moléculas biológicas
A tecnologia é derivada de um sistema de detecção e diagnóstico de câncer desenvolvido pela pesquisadora brasileira Lívia Eberlin na Universidade do Texas em Austin, baseado em espectrometria de massa – técnica que permite discriminar substâncias em amostras biológicas de acordo com a massa molecular.
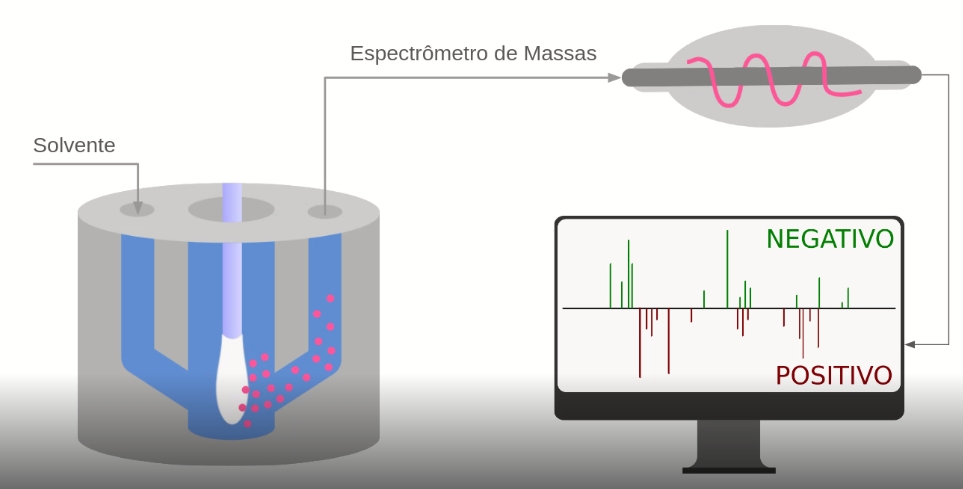
Batizado de MasSpec Pen, o método utiliza um dispositivo feito de plástico, na forma de uma caneta e esterilizável, para coletar moléculas biológicas da superfície de uma amostra de tecido.
A “tinta” da caneta é composta por água, utilizada como solvente para extrair moléculas de uma superfície de amostra de tecido, que são transportadas para um espectrômetro de massa para serem analisadas. Com base em algoritmos de aprendizado de máquina e modelos estatísticos o sistema é capaz de indicar se a amostra de tecido analisada contém células cancerosas.
“Basta a caneta só tocar o tecido para a água contida na ponta do dispositivo extrair as moléculas para análise”, afirma Eberlin.
Resultados de estudos clínicos iniciais indicaram que o sistema foi capaz de distinguir vários tecidos cancerígenos, incluindo tecidos tumorais de tireoide, mama, pulmão e ovário, de seus equivalentes normais com uma precisão geral de 96,3%.
“A ideia é que o sistema ajude os patologistas e cirurgiões a identificar mais rapidamente tecidos cancerígenos e tomar decisões mais precisas de tratamento”, diz Eberlin.
Detecção do SARS-CoV-2
Com o surgimento da pandemia de COVID-19, os pesquisadores tiveram a ideia de adaptar a tecnologia para detectar o SARS-CoV-2 diretamente nos esfregaços nasofaríngeos coletados por meio de swabs. Para isso, foram necessárias adaptações no design e nos solventes da caneta.
Como o dispositivo só toca uma superfície pequena de uma amostra de tecido e o material coletado por meio de swabs fica disperso, os pesquisadores decidiram inverter a caneta para que o cotonete nasal pudesse ser introduzido inteiramente em uma câmara – como o invólucro de uma caneta.
No interior da câmara, o swab entra em contato com uma pequena concentração de clorofórmio-metanol, usado como solvente para extrair as moléculas das secreções nasofaríngeas. As moléculas são sugadas por um orifício na câmara para um espectrômetro de massas para análise e identificação da presença de lipídeos que servem como marcadores para indicar se há ou não o vírus na amostra.
“Todo esse processo dura menos de um minuto, incluindo a análise. É um ciclo muito rápido, com etapas operacionais mínimas e sem a necessidade de uso de nenhum reagente especializado”, avalia Porcari.
Validação da tecnologia
Para validar o método, foram analisados inicialmente swabs nasofaríngeos de 244 pacientes atendidos no Hospital Bragantino e no Complexo Hospitalar Santa Casa Bragança Paulista, no interior paulista, no início da pandemia de COVID-19.
Com base nas análises, foi possível identificar os perfis lipídicos desses pacientes e gerar classificadores estatísticos para distinguir indivíduos sintomáticos positivos, sintomáticos negativos e assintomáticos negativos.
Os resultados do estudo indicaram que os perfis lipídicos detectados diretamente de esfregaços nasofaríngeos usando o novo método permitem a triagem rápida de pacientes com COVID-19.
“Esse novo método de análise de swabs pode ser adaptado para detecção de muitas outras infecções virais e bacterianas e para realização de exames como o papanicolau [para prevenção de câncer de colo de útero]”, afirma Eberlin.
Os pesquisadores pretendem realizar agora uma validação interlaboratorial em parceria com a Universidade Presbiteriana Mackenzie.
“A ideia é demonstrar com amostras independentes que esse novo método de análise é válido, independentemente dos laboratórios, equipamentos e dos analistas que realizam o teste”, explica Porcari
O artigo “Rapid screening of COVID-19 directly from clinical nasopharyngeal swabs using the MasSpec Pen” (DOI: 10.1021/acs.analchem.1c01937), de Kyana Y. Garza, Alex Ap. Rosini Silva, Jonas R. Rosa, Michael F. Keating, Sydney C. Povilaitis, Meredith Spradlin, Pedro H. Godoy Sanches, Alexandre Varão Moura, Junier Marrero Gutierrez, John Q. Lin, Jialing Zhang, Rachel J. DeHoog, Alena Bensussan, Sunil Badal, Danilo Cardoso de Oliveira, Pedro Henrique Dias Garcia, Lisamara Dias de Oliveira Negrini, Marcia Ap. Antonio, Thiago C. Canevari, Marcos N. Eberlin, Robert Tibshirani, Livia S. Eberlin e Andreia M. Porcari, pode ser lido na revista Analytical Chemistry em https://pubs.acs.org/doi/10.1021/acs.analchem.1c01937?ref=pdf&.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





