

Científicos brasileños desarrollaron un algoritmo con capacidad para identificar al protozoo Trypanosoma cruzi en fotos de muestras de sangre obtenidas con cámaras de teléfonos móviles. Este método de bajo costo aparece descrito en la revista PeerJ y puede reproducírselo (imágenes: archivo de los investigadores)
Científicos brasileños desarrollaron un algoritmo con capacidad para identificar al protozoo Trypanosoma cruzi en fotos de muestras de sangre obtenidas con cámaras de teléfonos móviles. Este método de bajo costo aparece descrito en la revista PeerJ y puede reproducírselo
Científicos brasileños desarrollaron un algoritmo con capacidad para identificar al protozoo Trypanosoma cruzi en fotos de muestras de sangre obtenidas con cámaras de teléfonos móviles. Este método de bajo costo aparece descrito en la revista PeerJ y puede reproducírselo
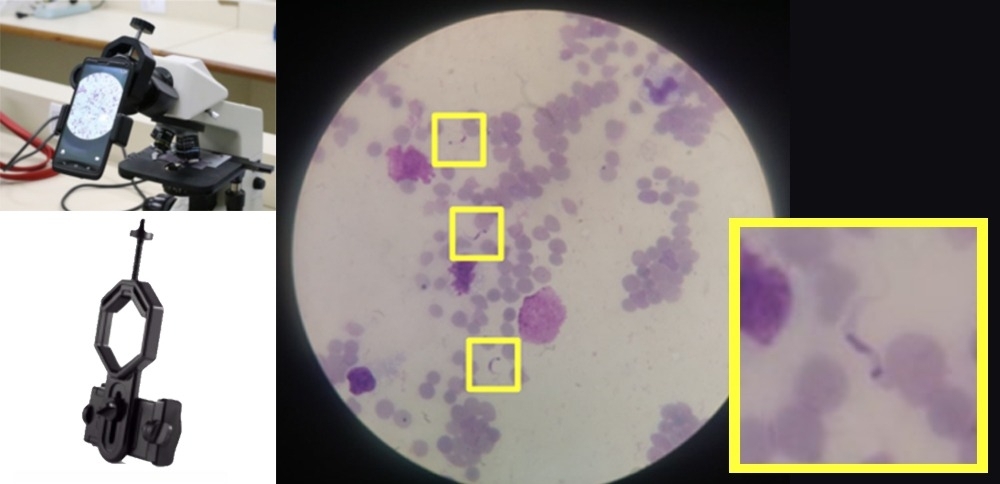
Científicos brasileños desarrollaron un algoritmo con capacidad para identificar al protozoo Trypanosoma cruzi en fotos de muestras de sangre obtenidas con cámaras de teléfonos móviles. Este método de bajo costo aparece descrito en la revista PeerJ y puede reproducírselo (imágenes: archivo de los investigadores)
Por Luciana Constantino | Agência FAPESP – Cuando el inmunólogo brasileño Helder Nakaya visitó el Instituto Evandro Chagas, en la ciudad de Belém (estado de Pará, Brasil), en 2017, había una conmoción porque uno de los mejores microscopistas de la entidad se iba a jubilar por entonces. De ese modo, gran parte de aquel conocimiento que permitía identificar rápidamente y con precisión cepas del protozoo Leishmania se perdería.
“Me intrigó ese desperdicio de toda esa capacidad acumulada durante décadas. Fue entonces que empezamos a investigar e intentar entrenar a la computadora con la sabiduría del profesional para actuar en la identificación de microorganismos y a bajo costo”, le explica Nakaya a Agência FAPESP.
Cinco años después, un grupo de investigadores, bajo la coordinación de Nakaya y del científico Mauro César Cafundó de Morais, publicó el resultado de un estudio demostrando que es posible utilizar la inteligencia artificial para detectar el Trypanosoma cruzi, el protozoario causante de la enfermedad de Chagas, en imágenes de muestras de sangre obtenidas con celulares y analizadas en microscopios ópticos.
El algoritmo que el grupo desarrolló se encuentra disponible en un artículo publicado en la revista científica PeerJ. La investigación contó con el apoyo de la FAPESP (proyectos 20/12017-9 y 15/22308-2) y congregó a profesionales de diversas áreas, desde la de biología hasta la de matemática y computación.
“Logramos un buen resultado de aprendizaje de la máquina. Con el algoritmo funcionado bien para la enfermedad de Chagas, podrá adaptárselo a otros propósitos que dependan de imágenes, tales como análisis de muestras de materia fecal, dermatología y colposcopía”, dice Nakaya, uno de los investigadores principales del Centro de Investigaciones en Enfermedades Inflamatorias (CRID), un Centro de Investigación, Innovación y Difusión (CEPID) de la FAPESP con sede en la Facultad de Medicina de Ribeirão Preto de la Universidad de São Paulo (FMRP-USP). Nakaya también desarrolla una investigación conjunta con Hospital Israelita Albert Einstein, la Plataforma Científica Pasteur-USP y el Instituto Todos pela Saúde.
Microscopistas capacitados efectúan una forma de diagnóstico de la enfermedad de Chagas: detectan los parásitos en muestras de sangre. Para ello se necesita un microscopio profesional, que puede acoplarse a una cámara de alta resolución, pero eso muchas veces vuelve a este método caro y de difícil acceso para personas de baja ingresos.
La enfermedad de Chagas, clasificada por la Organización Mundial de la Salud (OMS) como una de las enfermedades tropicales desatendidas (ETD), es considerada una condición infecciosa crónica, cuya prevención está relacionada con el modo de transmisión, es decir, al control del insecto conocido como vinchuca. Esto requiere de respuestas de las redes de atención de la salud.
La infección provocada por el Trypanosoma cruzi, que es endémica en 21 países del continente americano, afecta aproximadamente a 6 millones de personas, con una incidencia anual de 30 mil casos nuevos en la región, que provocan en promedio 14 mil muertes anuales. Asimismo, se estima que alrededor de 70 millones de personas viven en áreas de exposición a las vinchucas y corren riesgo de contraer a enfermedad.
En Brasil, aun cuando existe una tendencia de declive de las tasas de mortalidad, se ha registrado un promedio de 4.000 mil muertes por año ocasionadas por esta enfermedad durante la última década.
Para ‘enseñarle’ a la máquina
Los investigadores desarrollaron un abordaje de aprendizaje de la computadora basado en el denominado random forest (bosque aleatorio, en traducción libre), mediante la creación de un algoritmo para la detección y conteo de tripomastigotos del Trypanosoma cruzi en imágenes obtenidas con la cámara de un teléfono celular. Los tripomastigotos son la forma morfológica del protozoo presente en la fase extracelular y existente en la sangre de los pacientes con la enfermedad aguda.
Se analizaron micrografías de muestras de frotis de sangre registradas en imágenes con una resolución de 12 megapíxeles. Se extrajo un conjunto de parámetros morfométricos (forma y tamaño), color y mediciones de textura de 1.314 parásitos.
En esta etapa, los científicos João Santana Silva, Paola Minoprio y Ricardo Gazzinelli, especialistas en parásitos, ayudaron a “enseñarle” a la máquina a reconocerlos, especialmente al Trypanosoma. También colaboraron los investigadores de la USP Roberto Marcondes César Jr. y Luciano da Fontoura Costa, expertos en aprendizaje automático y procesamiento de imágenes.
Posteriormente, las muestras quedaron divididas en conjuntos de entrenamiento y pruebas, y entonces se las clasificó aplicando el algoritmo random forest. Los resultados fueron valores de precisión y sensibilidad considerados altos: se ubicaron en un 87,6 % y un 90,5 % respectivamente. Se analizó el área con la curva Característica Operativa del Receptor (curva ROC, por sus siglas en inglés), una representación gráfica que ilustra el desempeño o el rendimiento de un sistema clasificador binario a medida que su umbral de discriminación varía.
De este modo, el grupo logró automatizar el análisis de imágenes adquiridas con un dispositivo móvil, obteniendo así una alternativa con miras a achicar costos e incrementar la eficiencia en el empleo del microscopio óptico.
“La idea es generar imágenes y analizarlas en los microscopios que puedan ser enviados a lugares remotos de Brasil para que la propia aplicación indique si se trata o no del mal de Chagas. Por eso es importante contar también con un microscopio robusto y de bajo costo que pueda recolectar automáticamente las imágenes”, complementa Nakaya.
Según el investigador, la propuesta consiste en dejar el algoritmo abierto para que la comunidad científica aporte otros datos y otros recursos. Uno de los retos ahora reside en obtener microscopios de bajo costo, como el equipo producido en papel, inventado por los científicos Manu Prakash y Jim Cybulski en la Stanford University (Estados Unidos), pero que terminó no dando los resultados esperados al aplicárselo con los parásitos.
Puede leerse el artículo intitulado Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images en el siguiente enlace: peerj.com/articles/13470/.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





