

Mapa de interação entre proteínas de diferentes estudos (imagem: Daniel Martins-de-Souza)
Informações sobre conjunto de proteínas em uma amostra biológica tem ampla aplicação, sejam estudos de câncer, doenças mentais, neurodegenerativas ou cardiovasculares
Informações sobre conjunto de proteínas em uma amostra biológica tem ampla aplicação, sejam estudos de câncer, doenças mentais, neurodegenerativas ou cardiovasculares
Mapa de interação entre proteínas de diferentes estudos (imagem: Daniel Martins-de-Souza)
Ricardo Muniz | Agência FAPESP – Dificuldade comum em pesquisas que envolvem análises proteômicas é manejar e interpretar o imenso volume de informações obtidas. A técnica permite mapear simultaneamente milhares de proteínas e possibilita desvendar os intrincados mecanismos moleculares relacionados a doenças e condições biológicas – mas lidar com a complexidade de dados requer ajuda de diferentes algoritmos computacionais e tempo de aprendizado para operar cada um deles.
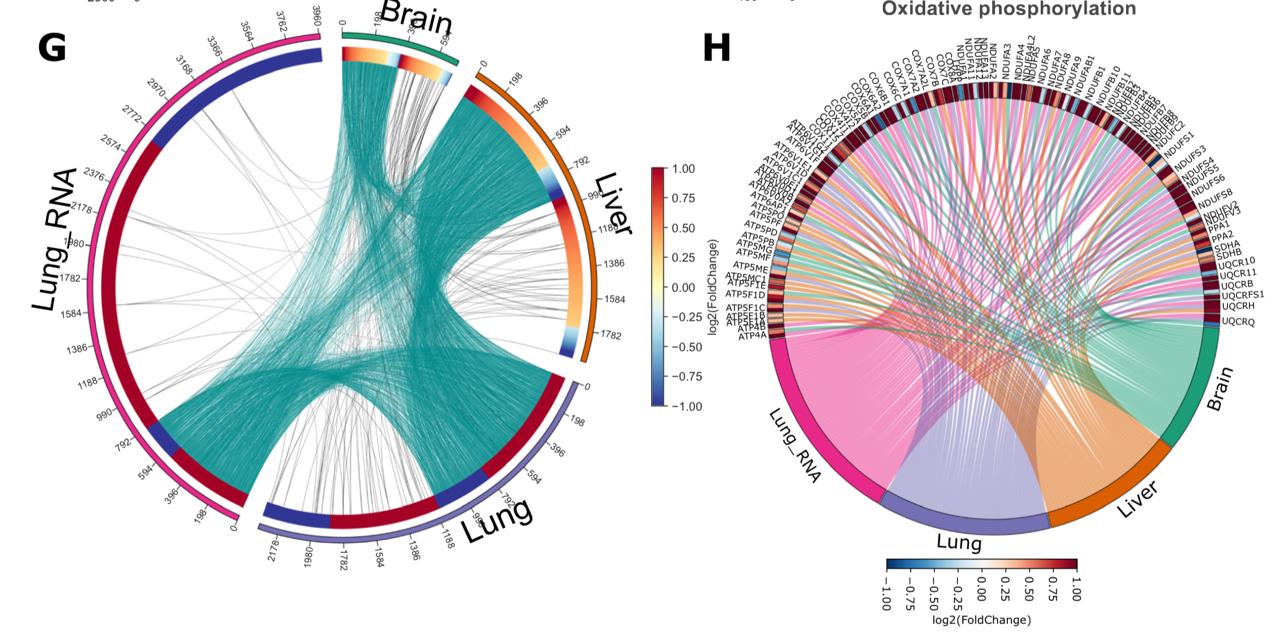
Artigo publicado na Nature Communications expõe uma solução que foi desenvolvida na Universidade Estadual de Campinas (Unicamp) em meio a uma série de estudos sobre proteínas relacionadas a casos de esquizofrenia, depressão e COVID-19. O trabalho é fruto da tese de doutorado de Guilherme Reis de Oliveira, bolsista da FAPESP.
“Trata-se de um programa computacional que consegue concatenar as informações do proteoma – o conjunto de proteínas produzidas por uma célula – e contextualizar como elas interagem entre si, além de mapear os processos biológicos nos quais estão envolvidas. É uma ferramenta que vem para suprir a necessidade de lidar com os dados em larga escala”, explica o orientador da tese, Daniel Martins-de-Souza, professor do Instituto de Biologia da Unicamp também apoiado pela FAPESP.
O chamado OmicScope é um sistema versátil, que ajuda a mapear moléculas não apenas relacionadas à esquizofrenia, sendo extensível praticamente a qualquer outra condição. “Com ele, qualquer proteoma pode ser analisado, seja para mamíferos ou até roedores, no caso de testes pré-clínicos, por conta dos mais de 220 bancos de dados integrados”, explica Martins-de-Souza. Assim, observa o pesquisador, a ferramenta pode servir para estudos sobre câncer, doenças mentais, neurodegenerativas ou cardiovasculares, entre muitas outras possibilidades.
“O OmicScope foi desenvolvido para as análises que sucedem os processos de identificação e quantificação de proteínas. É dividido em três componentes principais que realizam, respectivamente, proteômica diferencial, análise de enriquecimento e meta-análise. Cada módulo oferece seu próprio conjunto de funções, que incluem a geração de figuras e de redes, permitindo a análise dos dados”, explica Oliveira, na tese. Hospedado em servidor da Unicamp, pode ser acessado via web. Outro diferencial, apontam os autores, é que a ferramenta fornece uma variedade de arquivos de saída para integração com outros softwares, incluindo tabelas, imagens vetorizadas e arquivos de rede.
Embora as ferramentas já existentes suportem a análise de várias listas de genes, muitas não têm capacidade de conduzir uma análise ampla em múltiplos experimentos, nem de integrar dados com outras tecnologias, além de falhar com algumas informações importantes para determinados estudos. “O OmicScope foi projetado para lidar com vários formatos de informações. Realiza pré-processamento de dados – incluindo junção de réplicas, normalização, inserção de dados – e conduz análise proteômica diferencial para projetos experimentais estáticos e longitudinais”, afirmam os autores.
Imensidão de dados
A análise em larga escala de proteínas tem avançado significativamente a compreensão sobre mecanismos biológicos. “Hoje em dia é muito comum observarmos grandes volumes de dados sendo gerados pelas ciências ômicas, como a genômica. Essa vastidão de possibilidades facilita o nosso trabalho para que se consiga criar hipóteses e contar histórias biológicas sobre doenças que precisam ainda de esclarecimento”, destaca Martins-de-Souza.
Em laboratório, com Oliveira e os outros dois autores do artigo – Victor Carregari e Gabriel Souza –, Martins-de-Souza dedica-se a entender o proteoma do cérebro de pessoas que foram acometidas pela esquizofrenia, doença que afeta 1% da população mundial e quase 2,5 milhões de brasileiros.
Supervisionado por Martins-de-Souza e apoiado pela FAPESP, Carregari fez estudos de pós-doutorado na University of Southern Denmark, Dinamarca, onde investigou modificações pós-traducionais de proteínas em tecido cerebral de pessoas com esquizofrenia (projetos 23/04767-6 e 19/05155-9).
Um dos maiores obstáculos enfrentados pela maioria dos pacientes com essa doença é a baixa eficácia da medicação atual, decorrente das falhas de compreensão sobre o mecanismo biológico completo que leva à condição. “De forma geral, o que fazemos é mapear as proteínas de amostras de tecido de cérebros de pacientes com e sem esquizofrenia, e fazemos uma comparação”, conta Martins-de-Souza. Para isso, ele explica, precisam da ajuda de algoritmos computacionais como o OmicScope, que comportem a análise desses milhares de proteínas, cada uma com funções e participação em processos biológicos diferentes. “Quando você tem dezenas de milhares de proteínas para desvendar, precisa entender quais são os papéis que elas desempenham sozinhas e quais funcionam em conjunto, como interagem, como se regulam. É um volume de dados humanamente impossível de ser concatenado sozinho, por isso precisamos de eficientes algoritmos computacionais”, conclui.
O artigo OmicScope unravels systems-level insights from quantitative proteomics data pode ser lido em: www.nature.com/articles/s41467-024-50875-z#Ack1.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





