

Resultados de experimentos publicados na revista Oncotarget indicam que a inibição de um RNA não codificador denominado RMEL3 pode reduzir em até 95% a proliferação e a sobrevivência das células tumorais (Ilustração: Lucas Goedert e Cibele Cardoso)
Resultados de experimentos publicados na revista Oncotarget indicam que a inibição de um RNA não codificador denominado RMEL3 pode reduzir em até 95% a proliferação e a sobrevivência das células tumorais
Resultados de experimentos publicados na revista Oncotarget indicam que a inibição de um RNA não codificador denominado RMEL3 pode reduzir em até 95% a proliferação e a sobrevivência das células tumorais
Resultados de experimentos publicados na revista Oncotarget indicam que a inibição de um RNA não codificador denominado RMEL3 pode reduzir em até 95% a proliferação e a sobrevivência das células tumorais (Ilustração: Lucas Goedert e Cibele Cardoso)
Karina Toledo | Agência FAPESP – Estudo brasileiro divulgado na revista Oncotarget mostrou que a inibição de um RNA conhecido como RMEL3 pode reduzir em até 95% a viabilidade de células de melanoma em cultura.
Embora seja um RNA do tipo não codificador, ou seja, que não contém informações para a síntese de uma proteína, o RMEL3 parece modular – de forma ainda não totalmente compreendida – as principais vias de sinalização relacionadas à proliferação e à sobrevivência celular.
“Nossos estudos sugerem que RMEL3 está expresso na maioria dos casos de melanoma. Por outro lado, esse RNA é raramente encontrado em outros tipos de tumores ou mesmo em células sadias. Constitui, portanto um alvo terapêutico e diagnóstico bastante específico e promissor a ser explorado”, avaliou Enilza Espreafico, professora da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (FMRP-USP) e coordenadora da pesquisa apoiada pela FAPESP.
Os dados do artigo recentemente publicado são fruto de investigações feitas durante o doutorado dos bolsistas Lucas Goedert e Cristiano Gonçalves Pereira – com a colaboração de Cibele Cardoso e de outros pesquisadores no Brasil e no exterior.
A existência do RMEL3 e sua relação com o melanoma, porém, foram identificadas em um trabalho anterior do grupo de Espreafico – publicado em 2010 na revista PLoS One.
“Nossa intenção, na época, era identificar genes cuja expressão estivesse restrita a casos de melanoma. Usamos ferramentas de bioinformática para explorar bancos de dados criados a partir de projetos de sequenciamento de tumores”, contou a pesquisadora.
As primeiras análises revelaram a existência de 29 RNAs transcritos somente em células de melanoma. Três deles chamaram mais a atenção do grupo de Ribeirão Preto: o RMEL1, o RMEL2 e o RMEL3.
“Mostramos que esses três RNAs não codificadores de proteínas estão presentes tanto em linhagens celulares de melanoma quanto em amostras tumorais de paciente. O RMEL3 aparece até mesmo em lesões de pele consideradas pré-malignas”, contou Espreafico.
Os pesquisadores então passaram a investigar por meio de experimentos in vitro como a presença ou a ausência desses três RNAs alterava o fenótipo celular. Os primeiros resultados indicaram que a inibição de RMEL3 era o que resultava em maior queda na viabilidade das culturas de melanoma e, portanto, esse RNA se tornou o alvo preferencial da pesquisa.
Para silenciar o RMEL3 nas células em cultura, o grupo recorreu a um método conhecido como RNA de interferência, que consiste em usar pequenas moléculas de RNA não codificadoras de proteínas capazes de se ligar ao RNA transcrito do gene-alvo (no caso o RMEL3) e induzir sua degradação.
O efeito desse procedimento foi comparado pela equipe de Espreafico em cinco diferentes linhagens celulares. As três primeiras eram compostas por células de melanoma que apresentam uma mutação sabidamente associada ao câncer em um gene chamado BRAF. A quarta linhagem, também de melanoma, não tinha a mutação em BRAF. Já a terceira, considerada uma espécie de grupo controle, era uma linhagem de câncer de ovário que não expressava RMEL3 e nem apresentava a mutação em BRAF.
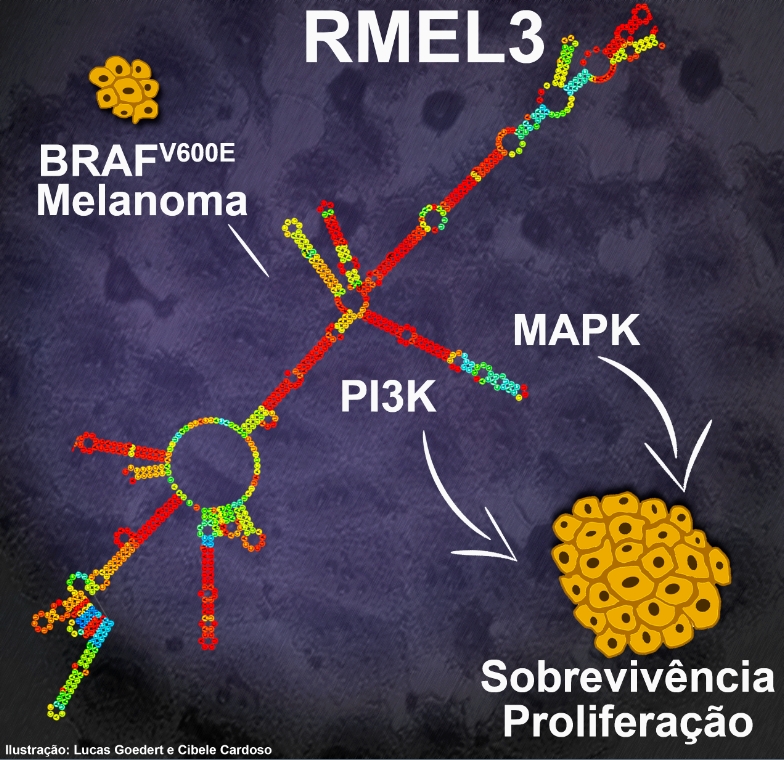
“BRAF é o principal proto-oncogene relacionado com o desenvolvimento de melanoma. Cerca de 60% dos casos desse tipo de câncer apresentam mutação no gene BRAF, que codifica uma proteína quinase iniciadora da via de sinalização MAPK – importante para ativar a proliferação celular” explicou a pesquisadora.
“Essa mutação altera apenas um código genético do gene BRAF, acarretando a troca de um aminoácido na cadeia polipeptídica. Isso é suficiente para criar a proteína oncogênica, conhecida como BRAF V600E. Em sua forma mutante, essa enzima é por si só ativa, o que faz com que a célula entre no ciclo de replicação mesmo sem receber qualquer sinal externo para proliferação”, acrescentou.
E foi justamente nas culturas de melanoma com a mutação BRAF V600E que a inibição de RMEL3 apresentou o efeito mais dramático, reduzindo em até 95% a sobrevivência e a proliferação celular. Na outra linhagem de melanoma, sem a mutação, a queda de viabilidade da cultura foi de aproximadamente 40%. Já na linhagem controle, o procedimento feito com o RNA de interferência não teve efeito algum, ou seja, as células continuaram proliferando normalmente.
O grupo ainda não sabe precisar qual é o papel desse RNA na célula ou por que ele está frequentemente presente nas células de melanoma. No entanto, já há indícios do que acontece na célula quando sua expressão é interrompida.
“Quando silenciamos o RMEL3, caem os níveis da proteína oncogênica BRAF e da quinase Akt/pAkt – proteína-chave da via de sinalização pró-sobrevivência celular conhecida como PI3K. O efeito oposto foi observado sobre a proteína PTEN, principal inibidor desta via”, contou Espreafico.
Ainda segundo a pesquisadora, foi observado aumento nos níveis de ACC-pS79, um substrato da enzima AMPK. Esta proteína, por sua vez, é um sensor da privação de nutrientes.
“Isso sugere que a falta de RMEL3 induz a um estado que mimetiza a privação de nutrientes, semelhante ao já reportado para a inibição farmacológica de BRAF V600E. Consistente com tais alterações, efetores do ciclo celular são alterados. Diminuem os níveis de ciclina B1, proteína importante para ativar o processo de mitose celular. Por outro lado, aumentam os níveis de proteínas que inibem o ciclo celular, como a p27 e a p21. Essas e outras alterações observadas são condizentes com o efeito que observamos nas células: aumento de morte celular e parada do ciclo celular”, contou Espreafico.
Para avançar na compreensão do papel de RMEL3 nas células, o grupo realiza atualmente novos experimentos in vitro nos quais a expressão desse RNA é artificialmente induzida tanto em células de melanoma como em células sadias, onde ele normalmente não estaria expresso. Os resultados devem ser divulgados em breve.
Potencial terapêutico
Com o intuito de descobrir o quão frequente é a expressão de RMEL3 nos casos de melanoma, o grupo avaliou quase 500 amostras de pacientes com esse tipo de câncer disponíveis no banco de dados do The Cancer Genome Atlas (TCGA), consórcio ligado ao National Cancer Institute, dos Estados Unidos, que reúne dados genômicos, epigenômicos e clínicos de pacientes de diversos países.
“Observamos que em mais de 90% das amostras de melanoma disponíveis o RMEL3 estava expresso – em maior ou menor grau. Isso, aliado ao fato de que ele não está presente nos demais tecidos sadios do organismo, o torna um alvo terapêutico muito interessante”, contou a pesquisadora.
Espreafico não descarta a possibilidade de usar o método de RNA de interferência como uma terapia. Alerta, porém, que antes é necessário vencer algumas barreiras técnicas.
“Seria preciso desenvolver uma tecnologia que aumentasse a estabilidade do RNA de interferência no organismo, pois são moléculas muito instáveis e se degradariam antes de alcançar o alvo. Já há grupos investigando algumas possibilidades, como o desenvolvimento de nanopartículas para proteger a molécula e aumentar sua concentração no sítio do tumor”, afirmou.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





