

Pesquisadores da USP e colaboradores desvendaram os mecanismos moleculares que tornam a cepa brasileira da bactéria E. coli produtora da toxina Shiga menos virulenta e patogênica do que a de outros países; resultados podem ser úteis para traçar estratégias de vigilância epidemiológica (Células epiteliais humanas infectadas pela STEC. As manchas vermelhas correspondem à toxina Shiga; imagem: S. Schuller / Wellcome Collection)
Pesquisadores da USP e colaboradores desvendaram os mecanismos moleculares que tornam a cepa brasileira da bactéria E. coli produtora da toxina Shiga menos virulenta e patogênica do que a de outros países; resultados podem ser úteis para traçar estratégias de vigilância epidemiológica
Pesquisadores da USP e colaboradores desvendaram os mecanismos moleculares que tornam a cepa brasileira da bactéria E. coli produtora da toxina Shiga menos virulenta e patogênica do que a de outros países; resultados podem ser úteis para traçar estratégias de vigilância epidemiológica
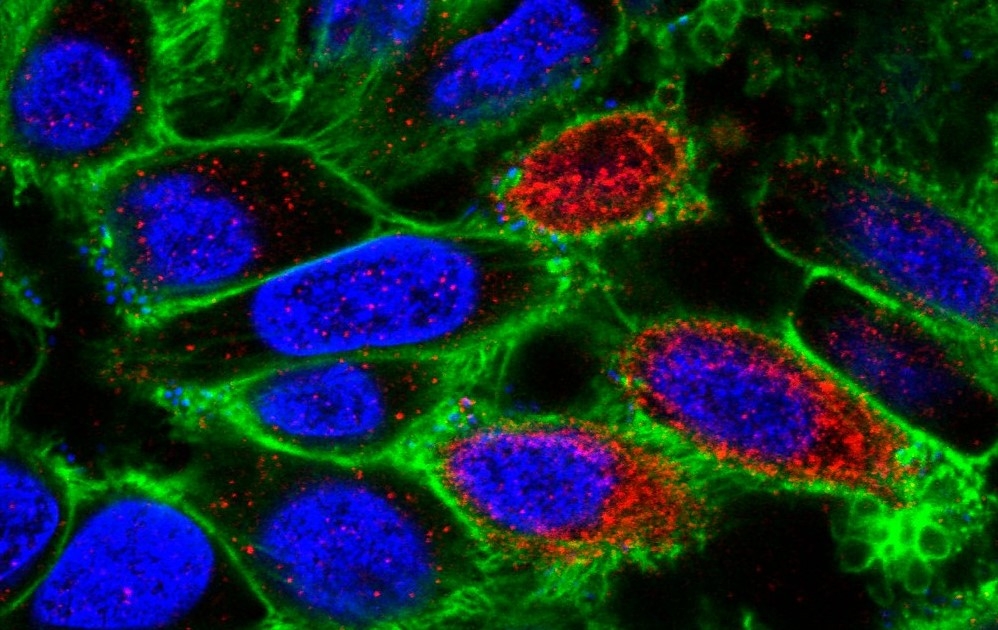
Pesquisadores da USP e colaboradores desvendaram os mecanismos moleculares que tornam a cepa brasileira da bactéria E. coli produtora da toxina Shiga menos virulenta e patogênica do que a de outros países; resultados podem ser úteis para traçar estratégias de vigilância epidemiológica (Células epiteliais humanas infectadas pela STEC. As manchas vermelhas correspondem à toxina Shiga; imagem: S. Schuller / Wellcome Collection)
Elton Alisson | Agência FAPESP – Entre as bactérias da espécie Escherichia coli, moradoras habituais do trato digestivo humano, existe um grupo capaz de produzir uma toxina chamada Shiga que tem intrigado os microbiologistas.
Em países como Austrália, Estados Unidos, Japão e Argentina, o consumo de carne bovina infectada por cepas dessa bactéria, conhecida como STEC (sigla em inglês para E. coli produtora de toxina Shiga), tem sido associado ao desenvolvimento de uma doença grave denominada síndrome hemolítico-urêmica (SHU), que causa insuficiência dos rins e acomete, principalmente, crianças com menos de cinco anos. No Brasil, por outro lado, um determinado sorotipo de STEC já foi encontrado na carne e nas fezes de bovinos – reservatório natural dessas bactérias –, mas nunca em pacientes diagnosticados com SHU.
Por meio de um estudo feito em colaboração com pesquisadores do Instituto Butantan e da Universidade Federal de São Paulo (Unifesp), cientistas da Universidade de São Paulo (USP) desvendaram, agora, alguns mecanismos moleculares que tornam a STEC “estrangeira” mais virulenta e patogênica que a cepa brasileira.
As descobertas, feitas no âmbito de um projeto apoiado pela FAPESP, foram publicadas e ganharam destaque de capa na revista Microorganisms.
“Os resultados do estudo podem ajudar a identificar novos marcadores moleculares de virulência e patogenicidade para bactérias STEC, que serão úteis para traçar estratégias de vigilância epidemiológica. O risco é a cepa brasileira sofrer alguma modificação genética que a torne capaz de também causar doença”, disse à Agência FAPESP Carlos Alberto Moreira Filho, professor do Departamento de Pediatria da Faculdade de Medicina da USP (FM-USP) e coordenador do projeto.
De acordo com Moreira Filho, a STEC causadora de SHU persiste mais tempo no intestino e libera paulatinamente a Shiga. A toxina atravessa a barreira intestinal, entra na corrente sanguínea e chega até os rins, onde causa lesão e, consequentemente, a insuficiência do órgão.
A fim de entender a diferença desse microrganismo para as cepas que não causam a doença, os pesquisadores fizeram um estudo, publicado em 2017 na revista PLOS ONE, em que compararam o genoma de uma cepa isolada de um paciente australiano diagnosticado com SHU, denominada EH41, e de uma cepa brasileira isolada de fezes bovinas, a Ec472/01.
Os resultados indicaram que na EH41 estão expressos 15 genes, entre eles o regulador de virulência dicA, que não estão ativos na cepa brasileira.
“Conseguimos, por meio desse estudo, esclarecer a diferença genética e genômica entre as cepas australiana e brasileira”, disse Moreira Filho.
A fim de analisar a resposta de células da camada superficial do intestino delgado às duas cepas, os pesquisadores fizeram, agora, um experimento com células humanas da linhagem Caco-2 cultivadas em laboratório.
“Essa linhagem de células mimetiza muito bem a camada de células epiteliais [enterócitos] do intestino delgado, à qual as bactérias STEC aderem para criar colônias”, explicou Moreira Filho.
As bactérias EH41 e a Ec472/01 foram colocadas sobre essas células durante três horas. Por meio de uma abordagem de biologia de sistemas, foram analisadas as alterações nos padrões globais de expressão gênica das culturas ao longo das três horas, em intervalos sequenciais de 7,5 minutos.
Já por meio de microscopia eletrônica de varredura foram avaliadas as alterações morfológicas nas células Caco-2 em intervalos de uma, duas e três horas.
Os resultados das análises indicaram que a cepa associada à SHU expressa um conjunto de genes de virulência que induz uma resposta diferente no enterócito humano e isso pode estar ligado à sua maior patogenicidade.
A ativação desses genes faz com que a cepa causadora de SHU impeça os enterócitos de expressarem determinadas reações de defesa, permitindo que o patógeno permaneça por mais tempo no intestino. Dessa forma, produz lentamente a toxina Shiga, que entra na corrente sanguínea e atinge os rins.
“Os enterócitos, que também são células de imunidade natural, falham na missão de deter a bactéria e permitem que ela persista no tecido e libere a toxina Shiga”, disse Moreira Filho.
Análises de redes
Para analisar a expressão e a interação dos genes dos enterócitos ao longo do tempo, induzida pela exposição às cepas das bactérias, foi usada uma metodologia de visualização em 3D de redes de coexpressão gênica por meio de um software desenvolvido por pesquisadores do Instituto de Física de São Carlos (IFSC) da USP. Também foram usados modelos matemáticos desenvolvidos pelo grupo do professor Roberto Marcondes César Junior, do Instituto de Matemática e Estatística (IME) da USP, por meio do projeto apoiado pela FAPESP.
Esses modelos matemáticos permitem identificar variações na topologia de uma rede – mudanças no nível hierárquico que os genes exercem em uma rede, em termos de influência sobre a expressão dos demais.
“Os algoritmos olham os genes funcionando e montam redes. Isso possibilita identificar agrupamentos ou módulos que explicam por que determinados genes estão conectados em uma situação e não em outras. Permite até mesmo simular o que a inativação de um determinado gene causaria em uma rede”, disse César Junior.
As análises mostraram que a bactéria associada à SHU causa uma variação abrupta da topologia das redes de coexpressão gênica das células Caco-2 ao longo das três horas de interação.
“A bactéria causa uma perda de conectividade entre os genes das células, que ficam com menos ligações”, disse Moreira Filho.
Já a análise dos grupos de genes mostraram que a bactéria causadora da doença induz a inflamação e a morte da célula Caco-2 logo na primeira hora de exposição, enquanto a cepa brasileira provocou menos alterações genéticas, principalmente na estrutura das células (o citoesqueleto) e uma resposta imune moderada.
A microscopia eletrônica, por sua vez, revelou que EH41, comparativamente à Ec472/01, causa nas células Caco-2 uma destruição muito mais intensa das microvilosidades, que são estruturas típicas de enterócitos sadios.
“Essas constatações ajudam a entender os mecanismos de indução de resposta imune que podem levar a condições graves, como a SHU", disse Moreira Filho.
Na avaliação do pesquisador, o estudo indica a necessidade de novas investigações sobre a influência de fatores ambientais na virulência das STEC, incluindo a passagem pelo trato digestivo de bovinos e humanos.
“Precisamos entender se a passagem dessa bactéria pelo sistema digestivo do gado brasileiro, que tem uma alimentação diferente, muda a expressão e desliga esses genes de virulência, por exemplo”, afirmou.
O estudo Dynamic gene network analysis of Caco-2 cell response to Shiga toxin-producing Escherichia coli-associated hemolytic-uremic syndrome (DOI: 10.3390/microorganisms7070195), de Silvia Y. Bando, Priscila Iamashita, Filipi N. Silva, Luciano da F. Costa, Cecilia M. Abe, Fernanda B. Bertonha, Beatriz E. C. Guth, André Fujita e Carlos A. Moreira-Filho, pode ser lido na revista Microorganisms em www.mdpi.com/2076-2607/7/7/195.
E o artigo A hemolytic-uremic syndrome-associated strain O113:H21.Shiga toxin-producing Escherichia coli specifically expresses a transcriptional module containing dicA and is related to gene network dysregulation in Caco-2 cells (DOI: 10.1371/journal.pone.0189613), de Silvia Yumi Bando, Priscila Iamashita, Beatriz E. Guth, Luis F. dos Santos, André Fujita, Cecilia M. Abe, Leandro R. Ferreira e Carlos Alberto Moreira-Filho, pode ser lido na revista PLOS ONE em journals.plos.org/plosone/article?id=10.1371/journal.pone.0189613.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





