

Enantiômeros são moléculas com a mesma estrutura e o mesmo número de átomos, porém, desviam o plano da luz polarizada para lados diferentes (imagem: Micael Cunha)
Pesquisadores brasileiros demonstram como a separação e a identificação de moléculas espelhadas são cruciais para o desenvolvimento de fármacos capazes de modular o processo de autofagia. Mau funcionamento desse mecanismo de limpeza das células está associado a doenças
Pesquisadores brasileiros demonstram como a separação e a identificação de moléculas espelhadas são cruciais para o desenvolvimento de fármacos capazes de modular o processo de autofagia. Mau funcionamento desse mecanismo de limpeza das células está associado a doenças
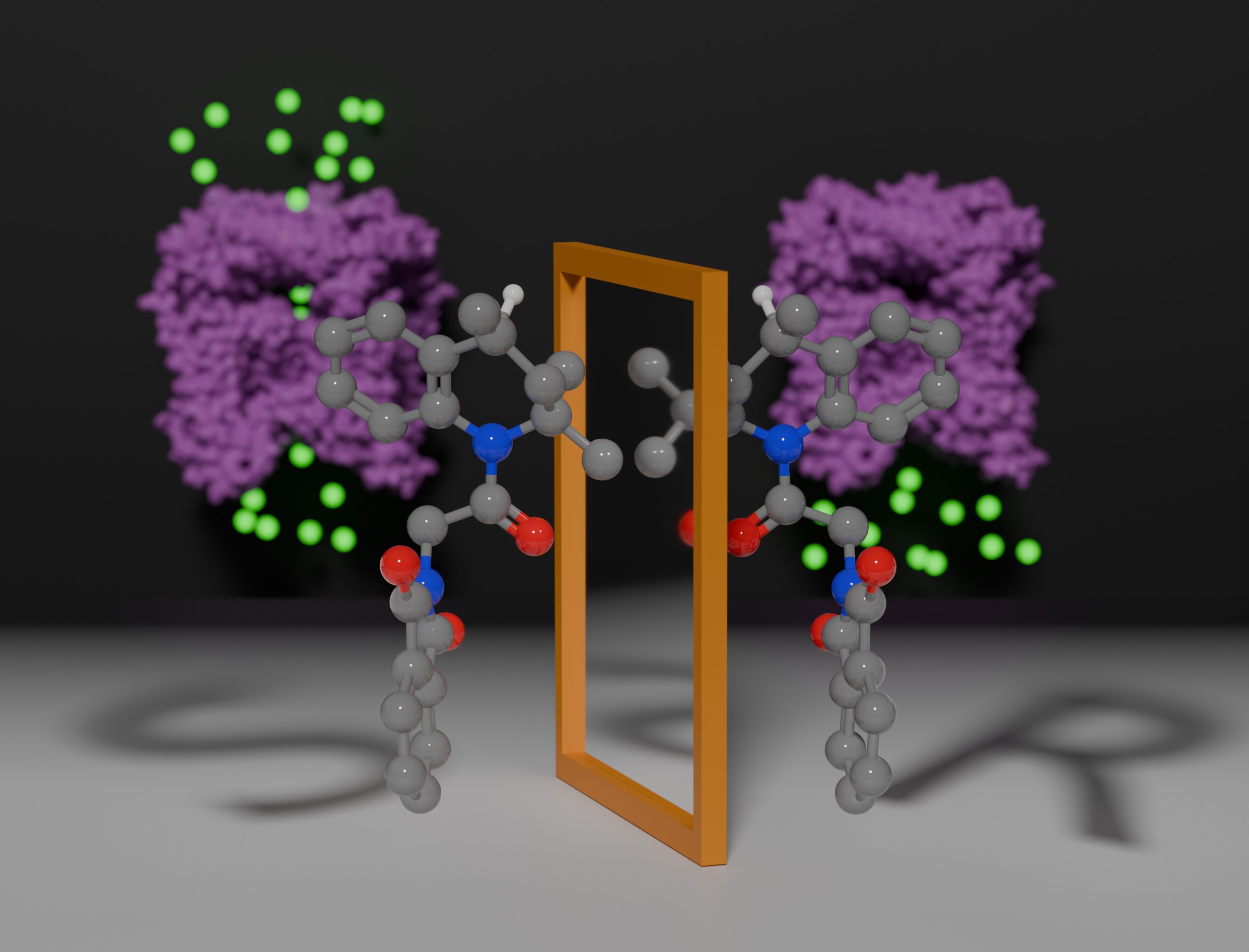
Enantiômeros são moléculas com a mesma estrutura e o mesmo número de átomos, porém, desviam o plano da luz polarizada para lados diferentes (imagem: Micael Cunha)
Agência FAPESP* – Uma etapa importante da pesquisa em química medicinal é encontrar moléculas que interajam diretamente com proteínas-alvo de forma potente e inequívoca, provocando uma cadeia de reações nas células. Este é um dos primeiros passos do processo de desenvolvimento de fármacos. Mas encontrar uma boa molécula não é suficiente, como destaca estudo recentemente publicado na revista ChemBioChem. É de fundamental importância verificar se ela é um enantiômero, ou seja, se existem outras versões espelhadas da molécula, seja no plano horizontal, vertical ou em ambos.
O artigo é assinado por pesquisadores das universidades Estadual de Campinas (Unicamp), Federal de São Paulo (Unifesp), Federal de São Carlos (UFSCar), Federal Fluminense (UFF) e do Instituto Federal de Educação, Ciência e Tecnologia de São Paulo (IFSP).
Para melhor entendê-lo, vale recordar alguns conceitos básicos de biologia celular. A célula tem várias organelas, entre elas os lisossomos. Eles têm formato de vesícula e são responsáveis por “reciclar” os componentes celulares. Ao encontrar resíduos no citoplasma, a vesícula os envolve e os lança para seu interior, onde serão digeridos por meio de um processo chamado de autofagia. Isso envolve a ativação e a desativação de proteínas que estão na membrana do lisossomo. O mau funcionamento dessas proteínas pode causar doenças como mucolipidose (grupo de distúrbios metabólicos hereditários caracterizados pelo acúmulo anormal de certas substâncias no meio intracelular, que causam uma série de consequências), tumores e maior suscetibilidade a infecções.
Quando o objetivo da pesquisa é buscar um composto capaz de interagir com a proteína existente na membrana do lisossomo, a TRPML1, o sucesso vai depender de qual versão da molécula está interagindo com esse alvo. Como demonstram os autores do artigo, essa informação otimiza a busca do melhor composto para estudar a proteína-alvo envolvida na reciclagem celular e cujo mau funcionamento está relacionado a doenças (leia mais em: agencia.fapesp.br/41711).
Para ter uma ideia de como intervir nesse mecanismo celular pode ser relevante, em novembro de 2022 a empresa Casma Therapeutics, que trabalha na descoberta de novos fármacos, recebeu um investimento de US$ 46 milhões para o desenvolvimento de medicamentos focados na autofagia. Já o grupo Merck, que atua no setor de ciência e tecnologia, investiu mais de US$ 1 bilhão na compra de duas empresas dedicadas ao desenvolvimento de ativadores da proteína TRPML1.
Os autores já sabiam que a molécula ML-SA1 (sigla em inglês para agonista sintético de mucolipina 1) era capaz de interagir com a proteína de membrana para ativá-la. “Notamos que apenas parte das moléculas conseguia interagir e ativar com sucesso a proteína-alvo”, explica Micael Cunha, bolsista de pós-doutorado da FAPESP. A resposta veio após os pesquisadores estabelecerem um protocolo de separação dos enantiômeros S e R da ML-SA1 e constatarem que apenas um deles tem efeito na proteína, ainda que ambos compartilhem a mesma estrutura e o mesmo número de átomos.
Tal informação é de extrema importância para os estudos baseados em alvo, pois revela que parte das moléculas usadas para ativar a TRPML1 está na verdade competindo pelo espaço de encaixe na proteína com as moléculas que realmente a ativam. Quando a forma S é testada isoladamente, a ativação é dez vezes maior do que o composto em mistura, enquanto a forma R não apresenta atividade. Segundo os resultados obtidos pelos pesquisadores, o enantiômero (R)-ML-SA1 se liga no mesmo local que o S, porém sem efeito. Seria como tentar vestir um calçado esquerdo no pé direito.
Separação das moléculas
Antes de testar as moléculas do tipo S e R isoladamente, foi preciso separá-las. Bruno Amaral, pesquisador do Centro de Química Medicinal (CQMED) da Unicamp, trabalhou na separação dos enantiômeros utilizados na pesquisa e explica que foi necessário estabelecer um novo protocolo, testando diferentes temperaturas, colunas de cromatografia e solventes.
A colaboração com a professora Quezia Cass, do Núcleo de Pesquisa em Cromatografia da UFSCar, possibilitou o desenvolvimento desse protocolo. “Conseguimos separar as moléculas em baixas temperaturas e usando etanol como solvente, o que torna o processo mais sustentável, pois não usa substâncias tóxicas ou que gerem descartes perigosos”, afirma Amaral.
Depois de separadas, os pesquisadores definiram qual era a versão S e R. “Quando eu consegui uma boa separação, só sabíamos que eram formas diferentes, mas não era possível identificar qual era a S e qual era a R por ser uma separação inédita e não haver informações suficientes na literatura para comparar”, explica. “Cruzamos dados teóricos e experimentais para identificar os enantiômeros e a colaboração do professor João Batista, da Unifesp, foi fundamental”, conta.
“As colaborações com diferentes universidades e laboratórios são fundamentais para conseguirmos avançar com a ciência de forma complementar”, comenta Katlin Massirer, pesquisadora do Centro de Biologia Molecular e Engenharia Genética (CBMEG-Unicamp) e coordenadora do estudo.
Apesar da boa permeabilidade e solubilidade adequada, a molécula ML-SA1(S) apresenta baixa estabilidade para ser usada como um medicamento para induzir a autofagia, suas aplicações atualmente se restringem ao campo das pesquisas. No entanto, os novos resultados apresentados sobre ela podem fornecer informações relevantes para o desenvolvimento de medicamentos estáveis, que sirvam para induzir o processo de autofagia em seres humanos. Fármacos para essa finalidade apresentam grande potencial terapêutico e econômico.
Iguais, mas diferentes
Enantiômeros são moléculas com a mesma estrutura e o mesmo número de átomos, porém, desviam o plano da luz polarizada para lados diferentes. Essa diferença é fundamental do ponto de vista químico e tem efeitos relevantes no funcionamento celular. Eles já são conhecidos pela ciência desde meados do século 19 e suas aplicações já são exploradas pela indústria farmacêutica e alimentícia.
O aspartame, um dos adoçantes alimentícios mais populares, pode apresentar quatro enantiômeros em sua conformação final (S,S), (S,R), (R,R) e (R,S), mas apenas a forma (S,S)-aspartame possui sabor adocicado. O limoneno é outro exemplo de como diferentes enantiômeros podem atuar. Este terpeno é naturalmente produzido por várias plantas, mas seu cheiro é determinado por sua conformação enantiomérica. Enquanto o (R)-limoneno possui odor de laranja, o (S)-limoneno apresenta odor de pinho.
Um trágico exemplo envolvendo enantiômeros foi o caso do medicamento talidomida. Em sua forma (R), a talidomida atua como um sedativo e por um tempo foi amplamente receitada para tratar enjoos de gestantes. O problema é que a forma (S) causa malformação e até mesmo a morte de fetos se ingerida durante a gestação, o que só foi descoberto depois de muitas vítimas já terem sofrido as consequências. O caso da talidomida demonstra a importância de se avaliar cuidadosamente as diferenças funcionais e toxicológicas dos enantiômeros presentes nos compostos antes de serem comercializados como medicamentos.
A pesquisa agora publicada recebeu financiamento da FAPESP por meio de outros três projetos (20/02006-0, 19/22319-5 e 14/50897-0). Também teve apoio do Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (Capes) e do Núcleo de Computação Científica da Universidade Estadual Paulista (NCC/Grid-Unesp).
O artigo (S)-ML-SA1 Activates Autophagy via TRPML1-TFEB Pathway pode ser lido em: https://chemistry-europe.onlinelibrary.wiley.com/doi/abs/10.1002/cbic.202400506.
* Com informações do CQMED.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





