

En la variante sudafricana B.1.351 (a la derecha), un anticuerpo se une menos a la proteína viral, formando un “valle” entre ambos, en un fenómeno que probablemente sucede debido a la glucosilación, indicada en la figura. En la P.1. (a la izquierda), la unión entre el anticuerpo y virus es mucho mayor (imagen: colección de los investigadores)
Una mutación en la proteína spike presente en ese linaje está asociada a un fenómeno bioquímico conocido con el nombre de glucosilación, que impide la unión de los anticuerpos. De confirmárselo mediante nuevos estudios, este descubrimiento puede orientar el desarrollo de vacunas más eficaces contra las nuevas cepas
Una mutación en la proteína spike presente en ese linaje está asociada a un fenómeno bioquímico conocido con el nombre de glucosilación, que impide la unión de los anticuerpos. De confirmárselo mediante nuevos estudios, este descubrimiento puede orientar el desarrollo de vacunas más eficaces contra las nuevas cepas
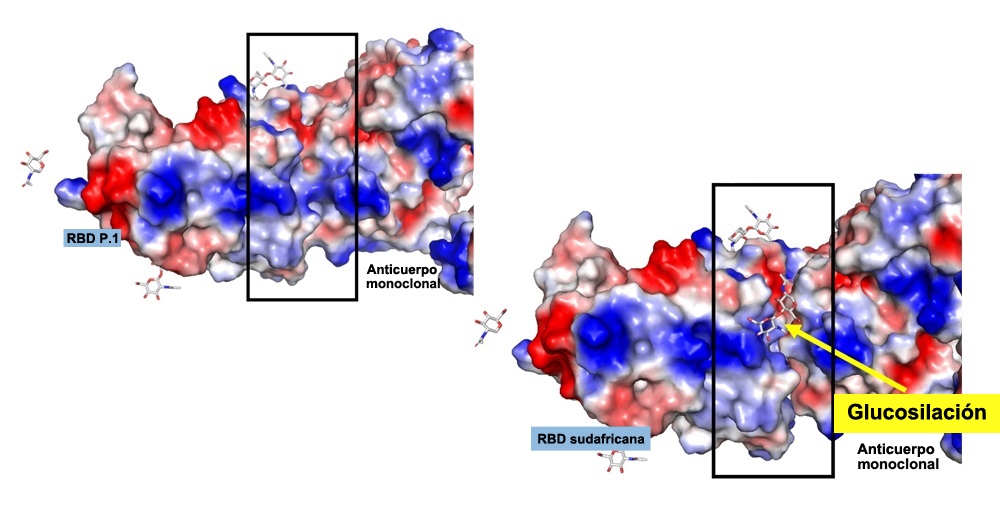
En la variante sudafricana B.1.351 (a la derecha), un anticuerpo se une menos a la proteína viral, formando un “valle” entre ambos, en un fenómeno que probablemente sucede debido a la glucosilación, indicada en la figura. En la P.1. (a la izquierda), la unión entre el anticuerpo y virus es mucho mayor (imagen: colección de los investigadores)
Por André Julião | Agência FAPESP – Un grupo de científicos encabezado por investigadores de la Facultad de Medicina de la Universidad de São Paulo (FM-USP), en Brasil, cree haber hallado el mecanismo que le permite a la variante sudafricana del SARS-CoV-2 –también conocida como B.1.351– escapar de los anticuerpos generados durante infecciones anteriores por la cepa ancestral del virus. Este descubrimiento, que aún debe confirmarse mediante nuevos experimentos, puede allanar el camino hacia el desarrollo de vacunas eficaces tanto contra la variante que surgió en Sudáfrica, presente en Brasil, como contra la originaria de Manaos (P.1.), como así también contra sus antecesoras.
Este estudio, publicado en la plataforma medRxiv, se encuentra en proceso de revisión por pares.
Mediante simulaciones computacionales, el grupo estudió la proteína clave del SARS-CoV-2, conocida como spike. Esta se encarga de unirse al receptor existente en las células humanas (la proteína ACE-2) para viabilizar la infección. Estos resultados sugieren que una de las mutaciones existentes en la punta de la proteína spike de la variante sudafricana –caracterizada por el cambio del aminoácido lisina por asparagina– puede redundar en el surgimiento de un fenómeno bioquímico conocido como glucosilación, que altera la fisonomía de la proteína viral e impide la unión de los anticuerpos. En tanto, en la variante P.1., la lisina es reemplazada por una treonina, que no sufre glucosilación.
“En el desarrollo de una vacuna, actualmente es necesario determinar qué será más eficaz contra el virus, incluyendo sus variantes. En el caso del SARS-CoV-2, de las tres mutaciones que existen en la P.1. y en la B.1.351, dos son exactamente iguales. Por ende, es posible que una vacuna enfocada en la variante sudafricana sea eficaz también contra la P.1. y contra el virus ancestral. Pero las vacunas contra estas dos últimas probablemente serán menos eficaces contra la variante sudafricana”, explica Keity Souza Santos, docente de la FM-USP y autora corresponsal del artículo.
Este trabajo es producto de un proyecto apoyado por la FAPESP y coordinado por Jorge Elias Kalil Filho, quien también suscribe el artículo. Kalil es docente de la FM-USP y coordina el Laboratorio de Inmunología del Instituto del Corazón (InCor) del Hospital de Clínicas, el complexo hospitalario administrado por la FM-USP.
El grupo liderado por Kalil trabaja en el desarrollo de una vacuna contra el COVID-19. Este proyecto también cuenta con el apoyo de la Fundación y con el la Finep, la Financiadora de Estudios y Proyectos, ligada al gobierno federal brasileño (lea más en: agencia.fapesp.br/32768/).
El blanco
“En el marco de trabajos anteriores de otros grupos, no se logró hallar región específica donde que los anticuerpos humanos se unen a la RBD [dominio de unión al receptor, por sus siglas en inglés], tal como se le denomina a la punta de la proteína spike que se encastra en las células humanas. Hasta ahora solamente se habían hecho inferencias. Utilizamos una técnica que nos permitió ubicar exactamente una región predominantemente reconocida, a la que denominamos inmunodominante. Es la misma donde está una de las mutaciones de las variantes de Manaos y de Sudáfrica”, comenta Souza Santos.
Tras identificar la región en la primera cepa del virus, el grupo de investigación –que congrega a científicos de universidades del estado de São Paulo (la USP, la Universidade Estadual Paulista y la Universidad Federal de São Paulo) y también de la Universidad de Salzburgo, en Austria– sometió la secuencia de aminoácidos a la acción del suero sanguíneo de 71 pacientes recuperados del COVID-19 en el Hospital de Clínicas al comienzo de la pandemia en Brasil. En el 68 % de las muestras, los anticuerpos presentes en el suero lograron unirse al péptido denominado P44, presente en la RBD de la proteína spike.
Para entender de qué manera transcurre la unión de los anticuerpos en esa área que los investigadores hallaron, se realizaron simulaciones computacionales. La información correspondiente a la RBD de ambas variantes y del virus ancestral se cruzó con la del anticuerpo monoclonal REGN10933, conocido por unirse a la región inmunodominante y, en la actualidad, en fase de ensayos clínicos para el tratamiento del COVID-19. La computadora efectúa lo que se denomina como predicción de neutralización, es decir, estima la capacidad de los anticuerpos para neutralizar el virus.
En las simulaciones, la predicción de neutralización fue completa contra el virus ancestral y un tanto menor para la variante P.1. Con todo, en la variante sudafricana se registró una disminución drástica en la predicción de neutralización, lo cual confirmó lo que se había apuntado en un artículo publicado en la revista Cell por científicos estadounidenses poco antes de la remisión del artículo de los brasileños.
Para el grupo encabezado por la USP, la unión no se concreta en la B.1.351 porque una de sus mutaciones consiste precisamente en el cambio del aminoácido lisina por la asparagina, que sufre el proceso de glucosilación. Esta alteración sería probablemente la responsable de la baja estimación de neutralización de la variante sudafricana a cargo de los anticuerpos generados a partir de la infección provocada por la cepa original del SARS-CoV-2. El fenómeno de la glucosilación ya se ha observado en el influenzavirus, el virus de la gripe, pero aún no se había hecho lo propio en el caso del SARS-CoV-2.
“En las variantes P.1. y B.1.351, la mutación de la RBD consiste en tan solo tres aminoácidos distintos con relación a la RBD del virus ancestral. Con todo, esta alteración parece ser suficiente como para que las variantes de Manaos y de Sudáfrica se vuelvan más transmisibles. Una vacuna que genere anticuerpos que ataquen a ambas mutaciones de las dos variantes en común, con el aminoácido glucosilado de la B.1.351, probablemente será más eficaz”, dice Souza Santos.
Para confirmar esta hipótesis, el grupo planifica ahora la realización de experimentos in vitro utilizando muestras del péptido P44 con la glucosilación en la asparagina. El objetivo es confirmar si los anticuerpos realmente no se unen a ese aminoácido cuando este es glucosilado. Asimismo, los investigadores obtuvieron suero de pacientes recuperados de la P.1. y pretenden confirmar si los anticuerpos de estos pacientes se unen efectivamente al péptido P44.
Puede leerse el artículo intitulado Immunodominant B cell epitope in SARS-CoV-2 RBD comprises a B.1.351 and P.1 mutation hotspot: implications for viral spread and antibody escape puede ser en el siguiente enlace: www.medrxiv.org/content/10.1101/2021.03.11.21253399v1.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





