

Científicos de Canadá y Brasil describen la utilización de 13 herramientas moleculares cuya función consiste en medir distintas vías de señalización intracelular y analizar los mecanismos de acción de nuevos fármacos (imagen: Science Signaling)
Científicos de Canadá y Brasil describen la utilización de 13 herramientas moleculares cuya función consiste en medir distintas vías de señalización intracelular y analizar los mecanismos de acción de nuevos fármacos
Científicos de Canadá y Brasil describen la utilización de 13 herramientas moleculares cuya función consiste en medir distintas vías de señalización intracelular y analizar los mecanismos de acción de nuevos fármacos
Científicos de Canadá y Brasil describen la utilización de 13 herramientas moleculares cuya función consiste en medir distintas vías de señalización intracelular y analizar los mecanismos de acción de nuevos fármacos (imagen: Science Signaling)
Por Maria Fernanda Ziegler | Agência FAPESP – Científicos canadienses en colaboración con pares brasileños desarrollaron 13 biosensores luminiscentes con el objetivo de verificar la eficacia de nuevos medicamentos in vitro.
Este estudio, que salió publicado en la revista Science Signaling, abre el camino hacia el análisis y la caracterización de nuevos fármacos, toda vez que los referidos biosensores tienen como base la actuación de receptores acoplados a proteínas G (del inglés G protein-coupled receptors, GPCR), localizadas en la membrana celular y cuya función consiste en efectuar la comunicación entre las células.
No es casual que el objeto de estudio lo constituyan los GPCRs. Se estima que entre un tercio y la mitad de todos los medicamentos comercializados actualmente tienen a esos receptores como blanco de acción.
“Estas proteínas son blancos de fármacos que se aplican en el tratamiento de una gran cantidad de afecciones: alergias, dolores, hipertensión y diabetes, entre otras. Cabe esperar que estos nuevos biosensores actúen también en el descubrimiento y en el desarrollo de nuevos fármacos destinado al tratamiento de una cantidad aún mayor aún de enfermedades”, dijo Claudio Miguel da Costa-Neto, docente del Departamento de Bioquímica e Inmunología de la Facultad de Medicina de Ribeirão Preto de la Universidad de São Paulo (FMRP-USP), en Brasil, y uno de los autores del artículo.
Da Costa-Neto explica que hace algunos años las pruebas in vitro para el desarrollo de nuevos medicamentos –se realizan antes que los ensayos en modelos animales y en humanos– mostraban únicamente si el compuesto activaba o bloqueaba una determinada respuesta celular.
“Haciendo una analogía: hace algunos años estos test se efectuaban como si existiera un interruptor que encendía o apagaba una lámpara. Recientemente nos dimos cuenta de que es posible analizar las distintas vías que puede activar a un receptor, como así también cuánto se activa una determinada vía de señalización. Por eso ahora la cosa ya no es tan sólo encender y apagar. Es como si contásemos con una sala con varias luces de LED, o con un dimmer que pueden mostrarnos cuáles y cuantas vías se activaron y, fundamentalmente, cómo se efectúa esa activación o el bloqueo. Nuestros biosensores, y los otros que están desarrollando otros grupos, suministran una respuesta más completa: algo así con un perfil de la señalización”, dijo.
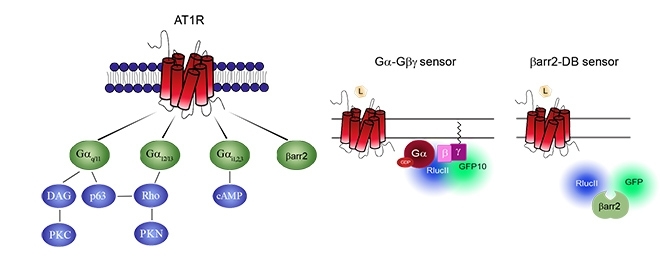
En el estudio, los investigadores desarrollaron herramientas destinadas a medir las distintas vías de señalización intracelular (biosensores de BRET, del inglés Bioluminescence Resonance Energy Transfer) y detectar la actuación de medicamentos en cultivos celulares. Sucede que las cascadas de señalización celular resultantes de las interacciones entre los receptores acoplados a proteínas G (GPCRs) y sus ligandos (proteínas) controlan una amplia variedad de procesos intracelulares, que así se convierten en los principales blancos con la mira puesta en el descubrimiento de nuevos fármacos.
Mediante el empleo de técnicas de ingeniería genética y biología molecular, el equipo internacional de científicos añadió enzimas fluorescentes y luminiscentes (tales como la luciferasa, la misma presente en las luciérnagas) en las proteínas que interactúan con los GPCRs.
“Cuando el fármaco activa al receptor –y por ende, las referidas proteínas presentes en el interior de las células entran en interacción con el receptor–, la luz que emite la luciferasa se traslada a la proteína fluorescente y ésta se activa. De este modo es como logramos medir con precisión los distintos niveles de acción de un fármaco o medicamento”, dijo Da Costa-Neto.
Este trabajo es el resultado de una amplia colaboración entre científicos de la FMRP-USP, de la Universidad de Montreal y de la McGill University, en Canadá. Y contó con cinco líneas de apoyo de la FAPESP: un Proyecto Temático, un proyecto seleccionado en el marco de una convocatoria del São Paulo Researchers in International Collaboration (SPRINT), una Beca de Investigación en el Exterior otorgada a la posdoctoranda Larissa de Bortoli y una beca doctoral concedida a Sarah Capelupe Simões; y también a través de la realización de la São Paulo School of Advanced Science on Medicines: from Target to Market, en marzo de 2018.
La validación
Los 13 biosensores se pusieron a pruebas en decenas de fármacos y también en diversos receptores mutantes, que simulaban polimorfismos correlacionados o no con enfermedades genéticas.
“Para validar herramientas tan importantes destinadas al descubrimiento de nuevos fármacos, los biosensores se testearon en distintos ligandos (fármacos) y también en receptores mutantes. Esto se hizo a los efectos de demostrar que es posible detectar un patrón distinto de señalización no sólo cuando se emplea un fármaco diferente, sino también cuando se altera el receptor (la proteína)”, dijo Da Costa-Neto.
De acuerdo con los investigadores, este estudio constituye un avance en la comprensión del mecanismo refinado de señalización, no sólo debido a la cantidad de biosensores desarrollados sino también a causa de la técnica que se empleó durante la investigación.
“Ya existían mecanismos como éstos que ahora mostramos, pero fuimos más a fondo en la comprensión. Por eso creemos que los nuevos biosensores tendrán un gran impacto sobre el desarrollo de nuevos medicamentos. Aparte de que los GPCRs constituyen blancos importantes, otro punto reside en que los biosensores han sido ostensivamente validados. Demostramos que funcionan, que responden bien y que son sumamente confiables para la caracterización de esas vías de señalización”, dijo Da Costa-Neto.
Además de la precisión de las herramientas desarrolladas, Da Costa-Neto destaca la creatividad como una de las bases de este estudio. “El desarrollo de proteínas mediante la aplicación de la ingeniería genética implica un gran conocimiento y mucha creatividad. Algunos de esos biosensores son verdaderos ‘frankensteins’, compuestos por la unión de diversas partes estructurales de distintas proteínas. Fue un estudio bastante creativo”, sostuvo el investigador.
Puede leerse el artículo intitulado Functional selectivity profiling of the angiotensin II type 1 receptor using pathway-wide BRET signaling sensors (doi: 10.1126/scisignal.aat1631), de Yoon Namkung, Christian LeGouill, Sahil Kumar, Yubo Cao, Larissa B. Teixeira, Viktoriya Lukasheva, Jenna Giubilaro, Sarah C. Simões, Jean-Michel Longpré, Dominic Devost, Terence E. Hébert, Graciela Piñeyro, Richard Leduc, Claudio M. Costa-Neto, Michel Bouvier y Stéphane A. Laporte. en el siguiente enlace: stke.sciencemag.org/content/11/559/eaat1631.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





