
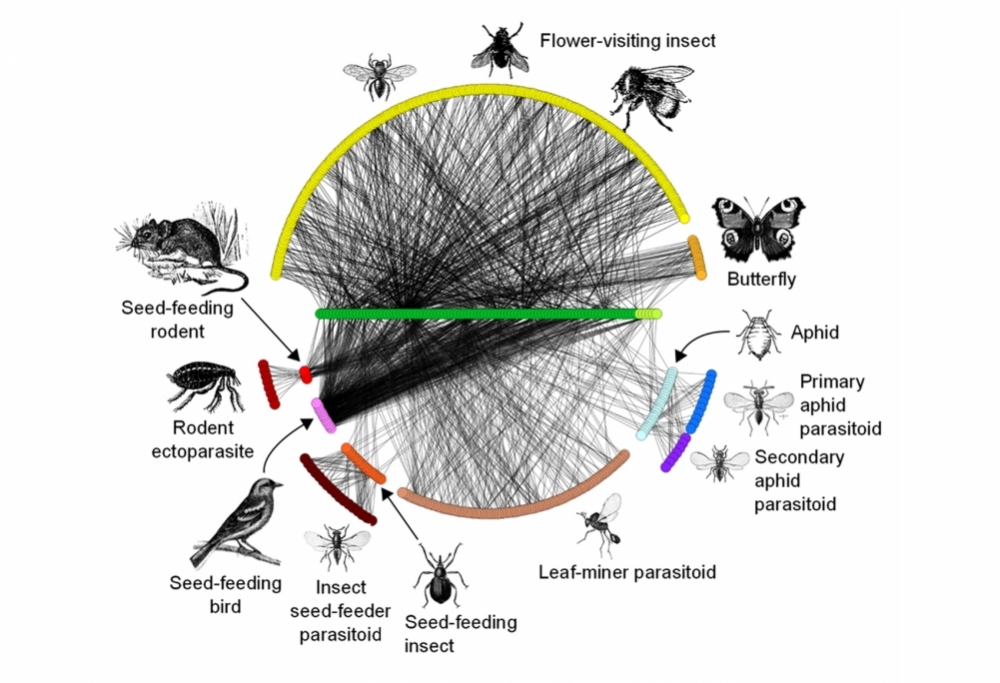
Una técnica conocida con el nombre de metabarcoding puede ayudar a los científicos a identificar especies de una determinada área y a entender cómo las mismas interactúan (imagen: Pocock, Evans & Memmott/ Science [2012] y Ecology Letters [2013])
Una técnica conocida con el nombre de metabarcoding puede ayudar a los científicos a identificar especies de una determinada área y a entender cómo las mismas interactúan
Una técnica conocida con el nombre de metabarcoding puede ayudar a los científicos a identificar especies de una determinada área y a entender cómo las mismas interactúan

Una técnica conocida con el nombre de metabarcoding puede ayudar a los científicos a identificar especies de una determinada área y a entender cómo las mismas interactúan (imagen: Pocock, Evans & Memmott/ Science [2012] y Ecology Letters [2013])
Por André Julião | Agência FAPESP – Al insertar una pequeña muestra de agua de río en un dispositivo menor que un smartphone, los científicos son capaces de determinar qué especies de peces, hongos, algas, invertebrados y bacterias viven en ese cuerpo de agua. Esto se ha vuelto posible gracias a la tecnología de secuenciación de ADN portátil que permite develar las interacciones entre éstas, además de saber qué especies son.
De acuerdo con Darren Evans, docente de la Newcastle University, en el Reino Unido, esta herramienta puede ayudar a gestionar mejor los ecosistemas e incluso a restaurar aquéllos que se han degradado.
Éste tema fue el tema que abordó el investigador durante su conferencia en la FAPESP Week London, un simposio realizado en la capital inglesa durante los días 11 y 12 de febrero de 2019.
“Lo que se hizo hasta ahora fue recolectar especies individuales y emplear la secuenciación para obtener barcodes [códigos de barras] de ADN únicos para esas especies”, dijo Evans, coordinador de un estudio realizado en colaboración con dos investigadores brasileños que contaron con el apoyo de la FAPESP (lea más en: agencia.fapesp.br/29669).
Mediante la aplicación del método conocido como código de barras de la vida o DNA barcoding, en lugar de efectuar la secuenciación de genes completos, se analiza únicamente un fragmento clave, que permite obtener información suficiente como para identificar a una especie.
“Toda esa información va a repositorios públicos y cualquiera puede obtenerla. Pero esto aún tarda mucho y tiene un costo alto. Lo que hicimos fue el metabarcoding, que se vale de una plataforma distinta con la cual podemos procesar quizá mil ejemplares en una ronda de secuenciación. Y en lugar de usar ese resultado sólo para crear barcodes de ADN de cada espécimen, también analizamos a sus parásitos, hongos y cualquier otro organismo asociado”, dijo.
Por ende, este nuevo método extiende la identificación de especies basada en ADN a comunidades de ejemplares pertenecientes a muchos grupos de especies con papeles distintos en el ecosistema. Los datos referentes a las interacciones de esos ejemplares permiten definir las redes existentes entre ellos y describir muy rápidamente la biodiversidad de un área.
“Esto revolucionará la forma de efectuar el monitoreo ambiental, porque se puede ir hasta un lugar y saber allí mismo qué organismos que viven en él y qué interacciones tienen entre ellos”, declaró Evans a Agência FAPESP.
Con información tan minuciosa, añadió, sería posible efectuar una mejor gestión de los ecosistemas e incluso recuperarlos, de ser necesario, mediante la aplicación de la denominada ingeniería de la biodiversidad.
En muchos hábitats, especies importantes se han extinguido dando espacio a otras similares, que pasaron a realizar la función de aquéllas que desaparecieron. Evans y sus colaboradores creen que es posible modelar ese escenario y determinar qué especies deberían estar en esa red.
“Tendríamos una lista con aquello que podría aumentar la resiliencia de esas redes. El reto reside en cómo poner en práctica estas ideas teóricas, restaurando sistemas naturales o sistemas agrícolas”, dijo.
Nitrógeno
La restauración de sistemas agrícolas degradados fue el tema de una conferencia durante la misma sesión de la FAPESP Week London. Este trabajo, realizado por científicos británicos y brasileños, se ha llevado adelante con productores rurales con el fin de enseñarles técnicas tendientes a equilibrar la cantidad de nitrógeno en el suelo, algo fundamental para la productividad de los cultivos.
“No siempre las técnicas que enseñamos tienen una buena aceptación entre los productores rurales, pues son más difíciles de aplicar o por otras razones. Éste es un dato que debemos tener en cuenta”, dijo Sacha Mooney, docente de la University of Nottingham, en el Reino Unido, durante su disertación.
Mooney presentó el proyecto NUCLEUS, un centro virtual para la mejora de la eficiencia del uso de nitrógeno entre productores rurales del Reino Unido y de Brasil. Este proyecto cuenta con el apoyo de la FAPESP y de agencias científicas de fomento británicas. En Brasil, el investigador responsable es Ciro Rosolem, docente de la Facultad de Ciencias Agronómicas de la Universidade Estadual Paulista (FCA-Unesp) en la localidad de Botucatu.
“Durante la primera etapa, les mostramos a los productores algunas sugerencias realmente exitosas en lo que hace al restablecimiento del nitrógeno en el suelo y al aumento de la productividad. La próxima etapa consiste en trabajar con quienes no han implementado ninguna de estas prácticas pues encontraron dificultades para hacer una inmersión en el problema y trabajar asociadamente”, dijo Mooney.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.




