

Forma dimérica da MPO mostrando o inibidor ZINC9089086, em amarelo, docado no sítio ativo da enzima MPO, em cinza (imagem: Isaac A. Matos/IQ-USP)
Grupo do Centro de Pesquisa em Processos Redox em Biomedicina da USP identificou novos inibidores da enzima mieloperoxidase, envolvida em vários processos inflamatórios. Compostos se mostraram eficazes em testes com animais
Grupo do Centro de Pesquisa em Processos Redox em Biomedicina da USP identificou novos inibidores da enzima mieloperoxidase, envolvida em vários processos inflamatórios. Compostos se mostraram eficazes em testes com animais
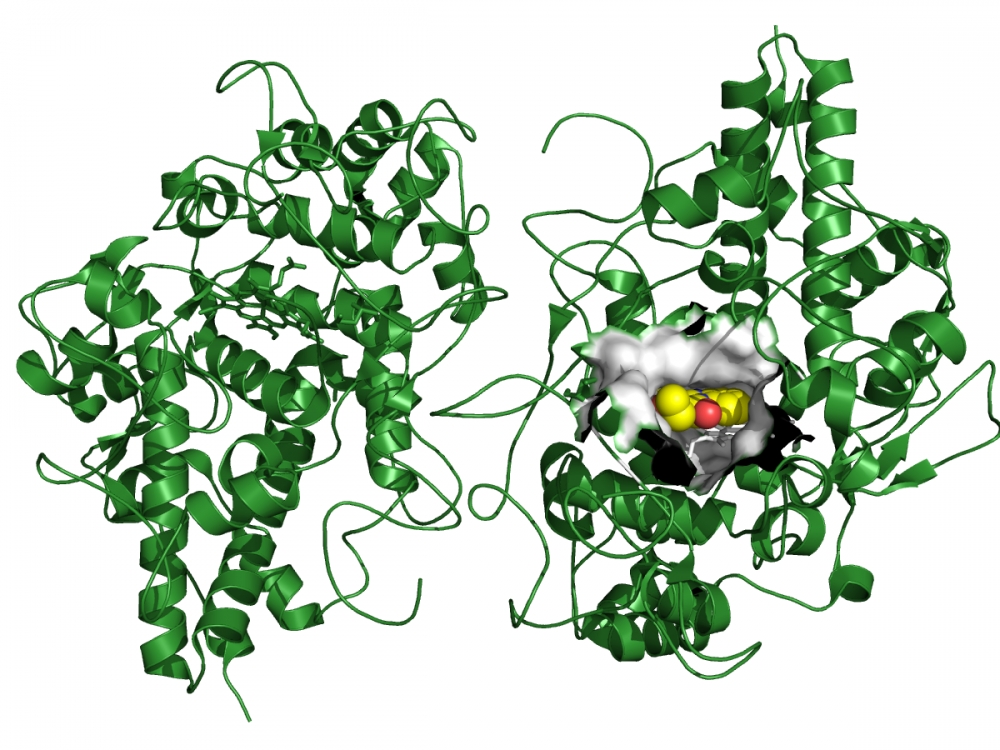
Forma dimérica da MPO mostrando o inibidor ZINC9089086, em amarelo, docado no sítio ativo da enzima MPO, em cinza (imagem: Isaac A. Matos/IQ-USP)
Agência FAPESP* – Cientistas do Centro de Pesquisa em Processos Redox em Biomedicina (Redoxoma) descobriram novos inibidores da enzima mieloperoxidase (MPO), envolvida em vários processos inflamatórios.
Os compostos se mostraram eficazes tanto em testes in vitro, feitos com linhagens de células humanas, como em um modelo animal de artrite gotosa – doença inflamatória, popularmente conhecida como gota, causada pela precipitação de cristais de urato nas articulações e que afeta cerca de 7% da população mundial.
Resultados da pesquisa foram publicados no Journal of Chemical Information and Modeling, periódico da American Chemical Society.
O novo método foi criado pelo pesquisador Isaac de Araújo Matos, durante seu doutorado. “A importância desse trabalho é o desenvolvimento de um método para a busca de inibidores da MPO com alta taxa de sucesso e diversidade química dos compostos descobertos. A ideia original foi usar uma metodologia genérica, que não se prendesse à estrutura do composto, mas trouxesse propriedades que aumentassem a probabilidade desse conjunto a ser testado de possuir verdadeiros inibidores da MPO”, conta o pesquisador.
O estudo foi realizado sob a supervisão da professora do Instituto de Química da Universidade de São Paulo (IQ-USP) Flavia Carla Meotti, integrante do Redoxoma – um Centro de Pesquisa, Inovação e Difusão (CEPID) financiado pela FAPESP.
Triagem virtual
Para criar o novo método, os pesquisadores analisaram propriedades moleculares genéricas, como massa molecular, polaridade e lipofilicidade (habilidade de um composto químico ser dissolvido em gorduras) de 143 inibidores já descritos. Em seguida, acrescentaram as regras de Lipinski e Veber, usadas para avaliar a biodisponibilidade oral dos compostos. Com isso, estabeleceram uma regra, que chamaram de regra MPO inibidor-símile.
Desse modo, fizeram a triagem virtual de mais de 35 milhões de moléculas disponíveis no banco ZINC12 na época e obtiveram 6.546 moléculas de interesse. O banco de dados ZINC é uma coleção de compostos químicos preparados especialmente para triagem virtual.
Como a regra inibidor-símile não é baseada em estrutura, os cientistas precisaram de uma metodologia para excluir os compostos que não podiam se ligar ao sítio ativo da enzima. “Usamos a triagem virtual baseada no receptor, a docagem molecular, na qual os 6.546 compostos foram encaixados dentro do sítio ativo da enzima. Foram usados vários programas, testados os melhores parâmetros para validação e a metade do conjunto foi analisada por inspeção visual”, conta Matos.
Nessa etapa, foram selecionados 242 compostos, que passaram por nova análise. Assim, eles identificaram dez candidatos, dos quais seis inibiam a MPO – uma taxa de sucesso de 60%. Os compostos apresentaram alta diversidade química e nenhum havia sido relatado anteriormente como inibidor de MPO. “Com isso a gente viu que a metodologia funcionava”, relata o pesquisador.
Matos explica que o método desenvolvido permite encontrar compostos com alta diversidade química. “E isso é fundamental, pois uma molécula às vezes inibe muito bem a enzima, mas é tóxica. Se você tem várias estruturas, pode selecionar a melhor. A gente quer diversidade química o máximo possível”, diz.
Em seguida, os pesquisadores voltaram a selecionar e testar 18 moléculas do mesmo conjunto de 242 compostos, das quais quatro foram testadas em um modelo animal de artrite gotosa.
“Vimos que, quando administrávamos os cristais de urato [causadores da inflamação] na pata, o edema triplicava e a atividade da MPO estava lá também. E, quando tratávamos com os inibidores da enzima, víamos uma redução significativa da inflamação”, conta Matos.
Todos os quatro compostos tiveram efeito antiedematoso, ou seja, diminuíram o inchaço da pata do animal, além de diminuir a atividade de peroxidase total e de citocinas pró-inflamatórias, quando administrados por via intraperitoneal, e três deles apresentaram o mesmo efeito quando administrados por via oral. Segundo o pesquisador, embora haja evidências da participação da MPO na gota, ainda não há estudos mostrando inibidores dessa enzima no tratamento da doença.
Dentre os compostos testados, um deles se mostrou mais potente como anti-inflamatório: a naftalamida. Esse composto tem alguma similaridade com o ABAH, ou 4-hidrazida do ácido benzoico, considerado um inibidor clássico da MPO, embora possua importantes diferenças estruturais e de mecanismo de ação.
“A naftalamida é uma molécula pequena e potente, que ainda não está pronta, mas pode facilmente ser otimizada. Como é uma nova classe química e farmacológica, nós fizemos uma patente das naftalamidas como inibidores da MPO para o desenvolvimento de drogas anti-inflamatórias”, explica o pesquisador.
O artigo Integration of an Inhibitor-like Rule and Structure-based Virtual Screening for the Discovery of Novel Myeloperoxidase Inhibitors pode ser acessado em: https://pubs.acs.org/doi/10.1021/acs.jcim.0c00813.
* Com informações da Assessoria de Comunicação do Redoxoma.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





