

Es un trabajo que salió publicado en la revista PLOS ONE, y sus ecuaciones sugieren que es posible estimar la variabilidad de la población vírica con base en datos epidemiológicos (imagen: archivo de los investigadores)
Es un trabajo que salió publicado en la revista PLOS ONE, y sus ecuaciones sugieren que es posible estimar la variabilidad de la población vírica con base en datos epidemiológicos
Es un trabajo que salió publicado en la revista PLOS ONE, y sus ecuaciones sugieren que es posible estimar la variabilidad de la población vírica con base en datos epidemiológicos
Es un trabajo que salió publicado en la revista PLOS ONE, y sus ecuaciones sugieren que es posible estimar la variabilidad de la población vírica con base en datos epidemiológicos (imagen: archivo de los investigadores)
Por José Tadeu Arantes | Agência FAPESP – Las mutaciones del SARS-CoV-2 constituyen uno de los temas más candentes del momento. Las nuevas variantes del virus están haciendo que la pandemia de COVID-19 recrudezca en lugares donde parecía estar controlada. Y pueden prolongar la fase crítica actual mucho más allá del tiempo esperado.
En el marco de un estudio realizado en el Instituto de Física Gleb Wataghin, de la Universidad de Campinas (IFGW-Unicamp), en Brasil, se modelaron las mutaciones que experimentó el SARS-CoV-2 durante su proceso de replicación y, por consiguiente, la evolución genética del virus en el transcurso de la pandemia. Y estos datos salieron publicados en la revista PLOS ONE.
En el artículo en cuestión, los autores emiten una voz de alerta que ya habían proferido otros científicos: las poblaciones que no están vacunándose y los grupos sociales que se rehúsan a ser vacunados favorecen el surgimiento de variantes. Y de no resolverse este problema con urgencia, la pandemia puede alcanzar un nuevo pico a escala global.
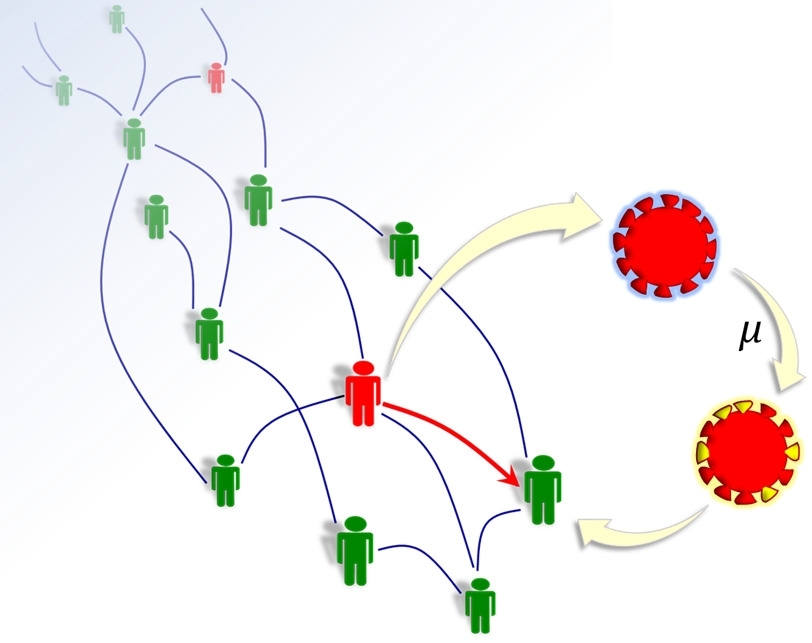
“Tal como se sabe, los virus son organismos muy sencillos, incapaces de reproducirse por sí mismos. Para poder replicar su ARN, deben utilizar las células del hospedante. Y al dañarlas, causan la enfermedad. Sucede que, durante el proceso de replicación, los errores de copia son inevitables. Los organismos más complejos poseen mecanismos de corrección de estos errores. Pero los virus no cuentan con ellos. En caso de que alguno de esos errores le brinde una ventaja al virus en términos de propagación, esa mutación pasará a tener importancia. Y eventualmente podrá incluso predominar. Si la propagación se concreta sin trabas, debido a la no vacunación, las mutaciones tienden a concretarse cada vez más y a propagarse por el planeta”, dice el físico Marcus de Aguiar, docente del IFGW-Unicamp y coordinador del estudio.
Al contrario de lo que dicen los negacionistas, no es la vacunación la que favorece las mutaciones, sino la falta de ella, según explica el investigador.
“Cuando se vacuna a gran parte de la población, el virus deja de circular. Y cuando circula menos, disminuye la tasa de reproducción viral. Y, por ende, disminuyen las chances de que aparezcan nuevas variantes.”
Los modelos tradicionales de epidemiología se enfocan en las cifras de personas infectadas, susceptibles y recuperadas en el transcurso del tiempo. Pero en el estudio en cuestión, el modelo incluyó la descripción del ARN del virus. “Es importante saber cuán distintos son los microorganismos en circulación en relación con los virus originales para entender la emergencia de nuevas variantes. Y también para estimar si, aunque ya se haya infectado con el virus original, una persona podrá infectarse nuevamente con una variante. Y para prever si el nuevo patógeno podrá o no escaparle a la acción de las vacunas proyectadas para el original”, explica De Aguiar.
Tal como sucede con cualquier modelo científico, el modelo que se desarrolló en el estudio constituye una aproximación idealmente simplificada a aquello que efectivamente sucede en la realidad. La base a partir de la cual se lo diseñó es el modelo del tipo SEIR, ya consagrado en epidemiología. La sigla SEIR está formada por las letras iniciales de cuatro palabras en inglés: susceptible (susceptible), exposed (expuesto), infectious (infectante) y recovered (recuperado). “Susceptible” es la persona que puede infectarse; “expuesta”, aquella que está infectada, pero que no se encuentra en estado infectante; “infectante”, la infectada e infectante, y “recuperada”, aquella que ya se ha recuperado de la enfermedad e, idealmente, no podría infectarse más.
“Para evitar una complejidad excesiva, que volvería al modelo matemáticamente inviable, consideramos que los individuos clasificados como ‘recuperados’ no pueden infectarse con ninguna variante que pueda surgir. También consideramos neutras a las mutaciones, es decir, que no dotan al virus de ninguna ventaja o desventaja adicional con relación al virus que le dio origen. Pero no es esto lo que sucede efectivamente en realidad. Así y todo, adoptamos estas simplificaciones para poder enfocarnos en nuestro objetivo, que era el de estudiar la acumulación de mutaciones víricas durante la pandemia y cuán distintos pueden volverse los virus”, aclara el investigador.
Para lograr este objetivo, se le agregó al modelo una descripción de los virus con base en su ARN, con 29.900 bases nitrogenadas, y una tasa de mutación 0,001 por base anual, datos obtenidos partiendo de la estructura y del comportamiento del SARS-CoV-2.
“Mientras que un individuo sigue infectado, el virus puede sufrir mutaciones y la persona puede transmitirlo. Calculamos la ‘distancia’ entre el virus original y la variante partiendo de la cantidad de bases nitrogenadas distintas que exhiben. Nuestras ecuaciones sugieren que es posible prever, con datos epidemiológicos [cantidad de personas susceptibles, infectadas y recuperadas], la variabilidad de la población viral [‘distancia promedio’ entre las secuencias de ARN], sin que sea necesario tener acceso a una enorme cantidad de datos genéticos”, dice De Aguiar.
Con el fin poner a prueba este modelo, los investigadores utilizaron las ecuaciones para demostrar, con base en datos de la epidemia provenientes de China, de comienzos de 2020, cómo sería la evolución de la “distancia genética promedio” entre los virus que hipotéticamente habrían surgido durante el referido período. Al comparar el resultado con las distancias calculadas con base en datos genéticos obtenidos localmente en idéntico período, el pronóstico mostró una buena concordancia con los datos reales.
“La propagación del virus a través de comunidades distintas [ciudades, países, etc.] puede llevar a secuencias bastante diferentes a la original, lo que eleva las probabilidades de infectarse nuevamente, lo cual depende en sobremanera de la conectividad entre esas comunidades. Cuanto menos conectadas están dos comunidades, mayor es la diferencia en el virus que una puede transmitirle a la otra. Esto incrementa las chances de que el virus circulante en una de las comunidades sea capaz de escaparle al control del sistema inmunitario de los individuos de la otra comunidad”, resume el investigador.
Y añade que “resulta importante remarcar que, para que se produzca una mutación efectiva del virus, que lo dote de ventajas o desventajas, es necesario que los defectos de replicación se materialicen en sitios específicos del ARN vírico. De este modo, las distancias genéticas grandes elevan las probabilidades de que se produzcan mutaciones importantes, pero no las aseguran. Y nuestras consideraciones se basan en esta perspectiva”.
Este estudio contó con el apoyo de la FAPESP en el marco de un Proyecto Temático, de una Ayuda de Investigación Regular concedida a De Aguiar, y de una Beca de Doctorado otorgada a Vitor Marquioni Monteiro, bajo la dirección de tesis de De Aguiar y autor principal del artículo.
Puede accederse a la lectura del artículo intitulado Modeling neutral viral mutations in the spread of SARS-CoV-2 epidemics en el siguiente enlace: journals.plos.org/plosone/article?id=10.1371/journal.pone.0255438.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.





