

Macrófagos derivados de medula óssea de camundongos das linhagens BALB/c (sensíveis à infecção) e C57BL/6 (resistentes à infecção) foram infectados com Leishmania amazonensis por 4 horas. O perfil dos transcritos dessas células mostrou que a infecção com o parasita apresenta um número maior de genes modulados em macrófagos de C57BL/6, o que pode indicar a razão desse camundongo estar mais preparado para resistir à infecção( Ilustração: Juliana Ide Aoki)
Estudo feito na USP mapeia o conjunto de genes que é ativado quando camundongos resistentes à Leishmania são infectados; moléculas que integram a via de sinalização responsável pelo combate ao parasita podem servir de biomarcadores para prognóstico da doença
Estudo feito na USP mapeia o conjunto de genes que é ativado quando camundongos resistentes à Leishmania são infectados; moléculas que integram a via de sinalização responsável pelo combate ao parasita podem servir de biomarcadores para prognóstico da doença
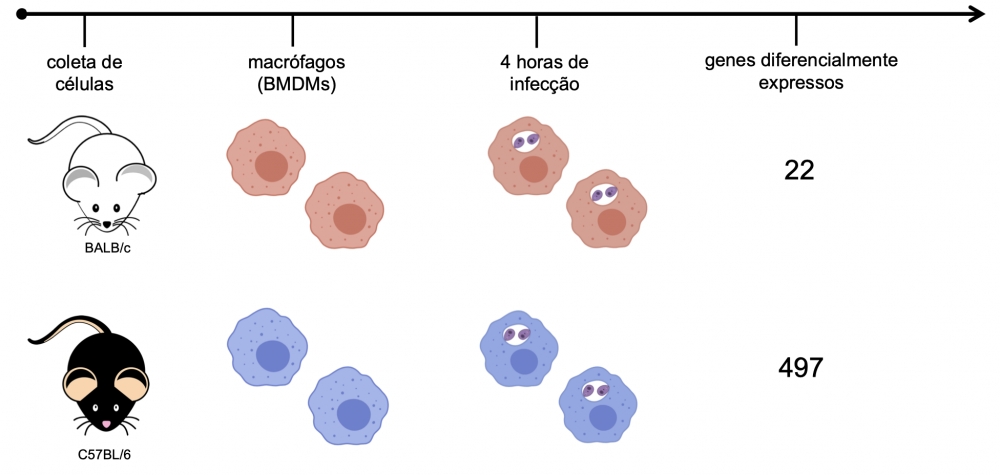
Macrófagos derivados de medula óssea de camundongos das linhagens BALB/c (sensíveis à infecção) e C57BL/6 (resistentes à infecção) foram infectados com Leishmania amazonensis por 4 horas. O perfil dos transcritos dessas células mostrou que a infecção com o parasita apresenta um número maior de genes modulados em macrófagos de C57BL/6, o que pode indicar a razão desse camundongo estar mais preparado para resistir à infecção( Ilustração: Juliana Ide Aoki)
Janaína Simões | Agência FAPESP – Em artigo publicado na revista Scientific Reports, pesquisadores da Universidade de São Paulo (USP) descreveram, em camundongos, como fatores genéticos podem determinar se um indivíduo é suscetível ou resistente à leishmaniose. Segundo os autores, os resultados podem ajudar a entender por que também entre os humanos apenas parte dos infectados desenvolve a doença.
A leishmaniose é causada por protozoários do gênero Leishmania que, dependendo da espécie, causam ulcerações na pele (leishmaniose cutânea) ou lesões em órgãos como fígado e baço (leishmaniose visceral). Os parasitas são transmitidos para humanos e outros mamíferos por meio da picada de insetos. Não há vacina disponível e o tratamento é longo, caro e complicado.
“Ao observar a regulação dos genes em nosso modelo de estudo, procuramos entender qual é a estratégia adotada por um organismo resistente à infecção e identificar que diferenças existem em relação àquelas do organismo sensível”, conta Lucile Maria Floeter-Winter, coordenadora da pesquisa no Laboratório de Fisiologia do Instituto de Biociências (IB-USP).
A investigação foi conduzida no âmbito de dois Projetos Temáticos apoiados pela FAPESP: “Estudos bioquímicos, moleculares e funcionais da relação Leishmania-macrófago" e “A relação Leishmania-hospedeiro sob a ótica das ‘ômicas.”
Os testes foram feitos com duas linhagens de camundongos: a BALB/c, naturalmente sensível ao parasita, e a C57BL/6, naturalmente resistente. Ambas foram infectadas pela espécie Leishmania amazonensis, causadora da forma cutânea da doença. Na avaliação dos pesquisadores, prognósticos da doença podem ser estabelecidos quando tivermos marcadores moleculares que indiquem a capacidade de resistir ou não à infecção.
Mapeamento da expressão gênica
Os parasitas do gênero Leishmania têm como estratégia infectar justamente as células que deveriam combatê-los: os macrófagos. Nessas células eles se reproduzem para depois infectar outros macrófagos do organismo hospedeiro.
Quando o parasita infecta o macrófago, duas coisas podem acontecer: ou ele arrebenta, liberando os parasitas que então infectam outros macrófagos ao redor – caso em que se considera a infecção como estabelecida – ou o parasita é morto pelo macrófago e a doença não avança.
Para criar um modelo experimental da infecção, os pesquisadores retiraram células da medula óssea dos camundongos, diferenciaram essas células em macrófagos e as infectaram com o parasita. Foram consideradas no estudo apenas as primeiras quatro horas depois da infecção, quando os macrófagos infectados foram usados para extração de RNA e seu sequenciamento. O mapeamento da expressão dos genes, obtido pela análise do transcriptoma de ambas as linhagens de camundongos, permitiu verificar quais genes estavam sendo expressos durante as primeiras quatro horas de infecção em cada grupo.
“Focamos nossas atenções no começo da infecção, quando os mecanismos de resposta são disparados”, explica a pesquisadora do IB-USP Juliana Ide Aoki, autora principal do artigo e bolsista de pós-doutorado da FAPESP.
“Ao ser infectado, o organismo dispara um aviso para que o macrófago produza um conjunto de moléculas responsáveis pela resposta à infecção. Quando analisamos o transcriptoma, conseguimos ver quais segmentos do genoma entenderam esse sinal e efetuaram uma resposta para combater o parasita”, diz Floeter-Winter.
No conjunto dos genes expressos dos macrófagos estudados em cultura, foi identificado um total de 12.641 genes. Contudo, no BALB/c (sensível), apenas um conjunto de 22 genes teve a expressão modificada, como resposta de combate ao parasita. Número bem menor do que os 497 observados nos camundongos C57BL/6 (resistente).
“Esse é o grande achado da pesquisa. Temos um organismo que aciona pouquíssimos genes e não é capaz de conter a infecção e, de outro lado, um que aciona muitos genes que induzem a produção de moléculas para controlar a infecção e consegue atingir esse objetivo”, conta Floeter-Winter.
“Nossos resultados mostram que realmente o desenvolvimento da doença depende da genética do hospedeiro, não só do parasita, e pode explicar por que existem pacientes que desenvolvem a infecção enquanto outros se mostram resistentes”, diz.
De acordo com a pesquisadora, os camundongos foram tratados da mesma forma e receberam a mesma alimentação, o que descarta a influência de fatores ambientais nos resultados. Permanece como tema para pesquisas posteriores desvendar por que os animais C57BL/6 ativam mais genes para combater a infecção.
A determinação de moléculas presentes em um organismo infectado que é capaz de controlar a infecção permite sugerir marcadores para avaliação e prognóstico da doença em pacientes humanos. “Seria possível, por exemplo, ver que moléculas o paciente infectado está expressando e estabelecer um prognóstico se ele irá desenvolver uma doença mais longa, se vai desenvolver uma infecção com potencial mais drástico, ou se ele produz moléculas que combatem a infecção”, diz a pesquisadora.
Além disso, a contribuição do estudo pode ser extrapolada para outros aspectos dessa doença. “Ao entendermos como as Leishmanias conseguem estabelecer a infecção, descrevemos mecanismos de resposta que podem ser utilizados em outras doenças infecciosas, fornecendo informações a outros pesquisadores na investigação em outros modelos”, finaliza.
O estudo faz parte de um conjunto de pesquisas coordenadas por Floeter-Winter que busca entender a relação entre hospedeiro e parasita, como a que identificou potenciais alvos para o tratamento da leishmaniose (leia mais em: agencia.fapesp.br/25961).
Além de Aoki e Floeter-Winter, assinam o artigo Sandra Marcia Muxel e Ricardo Andrade Zampieri, do IB-USP, e Audun Helge Nerland, da Universidade de Bergen e Karl Erik Müller, do Drammen Hospital, ambos localizados na Noruega.
O artigo Differential immune response modulation in early Leishmania amazonensis infection of BALB/c and C57BL/6 macrophages based on transcriptome profiles pode ser lido em: www.nature.com/articles/s41598-019-56305-1.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





