

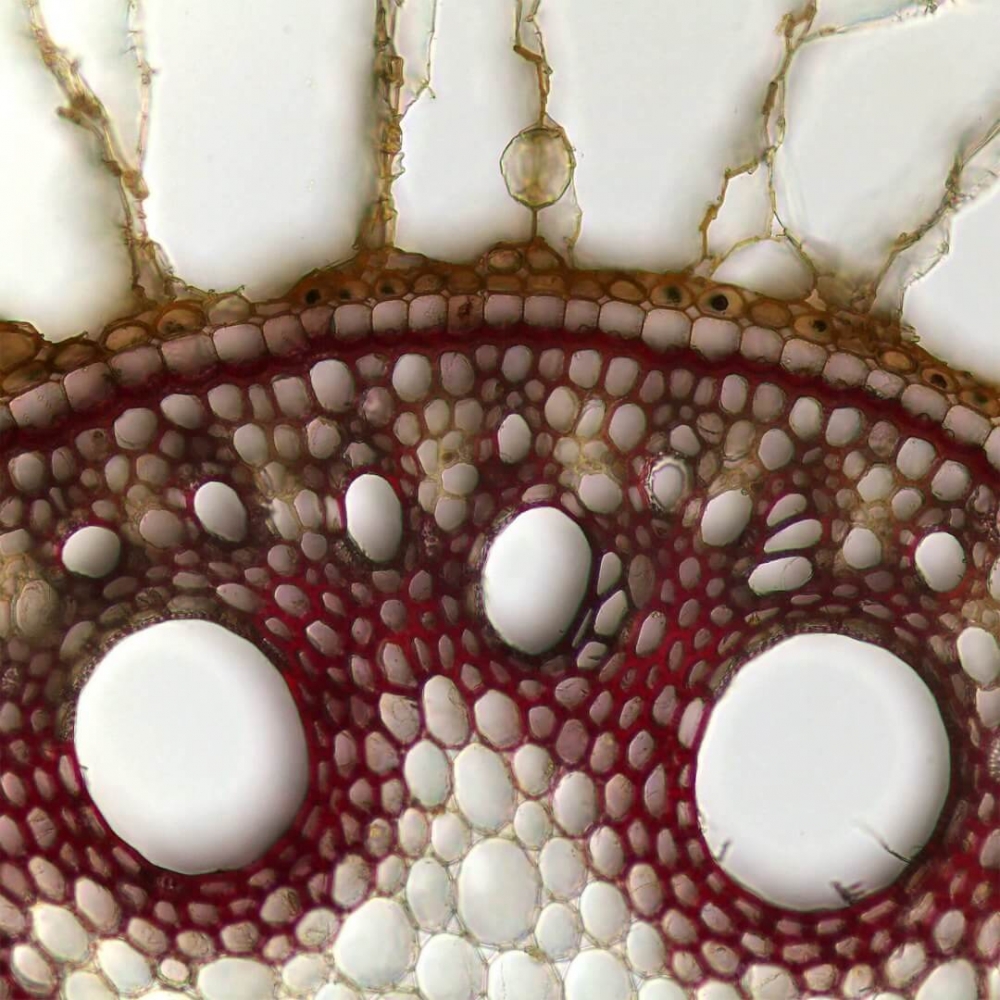
Descrição de genes envolvidos na separação celular que ocorre na raiz da cana-de-açúcar possibilita o desenvolvimento de variedades com a parede celular amolecida, como a do mamão, favorecendo o etanol de segunda geração (imagem: microscopia do aerênquima da raiz de cana / Débora Chaves Coelho Leite)
Descrição de genes envolvidos na separação celular que ocorre na raiz da cana-de-açúcar possibilita o desenvolvimento de variedades com a parede celular amolecida, como a do mamão, favorecendo o etanol de segunda geração
Descrição de genes envolvidos na separação celular que ocorre na raiz da cana-de-açúcar possibilita o desenvolvimento de variedades com a parede celular amolecida, como a do mamão, favorecendo o etanol de segunda geração
Descrição de genes envolvidos na separação celular que ocorre na raiz da cana-de-açúcar possibilita o desenvolvimento de variedades com a parede celular amolecida, como a do mamão, favorecendo o etanol de segunda geração (imagem: microscopia do aerênquima da raiz de cana / Débora Chaves Coelho Leite)
Elton Alisson | Agência FAPESP – Durante o período de amadurecimento do mamão (Carica papaya), as células da parede celular do fruto se separam, tornando o tecido mais amolecido e de fácil digestão. Esse processo permite a disponibilização de conteúdos celulares e facilita a extração do açúcar (sacarose) da fruta ao ser ingerida.
Recentemente, constatou-se que a raiz da cana-de-açúcar realiza um processo similar. Durante seu desenvolvimento, as paredes celulares são modificadas e formam-se espaços preenchidos de ar (chamados de aerênquimas) que separam as células.
“Os aerênquimas são muito comuns em plantas alagadas, como o arroz, pois favorece a sustentação ou a flutuação na água, a chegada de oxigênio e a retirada de gás carbônico das partes submersas do vegetal”, disse Marcos Buckeridge, professor do Instituto de Biociências da Universidade de São Paulo (IB-USP).
Nos últimos anos, Buckeridge e colaboradores têm se dedicado a estudar os genes envolvidos na separação celular na raiz da cana a fim de desenvolver variedades transgênicas da planta que permitam que esse processo ocorra em outras partes, como no colmo, onde se acumulam biomassa e sacarose.
Dessa forma, seria possível cultivar variedades de cana com a parede celular amolecida como a de um mamão – a chamada “cana papaia”. E, com isso, facilitar a degradação da parede celular e viabilizar a produção em larga escala de bioetanol de segunda geração (obtido a partir da biomassa).
Agora, um grupo de pesquisadores vinculados ao Instituto Nacional de Ciência e Tecnologia do Bioetanol – um dos INCTs apoiados pela FAPESP em São Paulo em parceria com o Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) – deram um importante passo nesse sentido.
Eles conseguiram descrever, em parceria com colegas de outras universidades e instituições de pesquisa no Brasil e no exterior, as primeiras sequências de genes envolvidos na separação celular na raiz da cana e elucidar suas funções nesse processo. Os resultados do estudo, foram publicados no Journal of Experimental Botany.
“Se conseguirmos promover o efeito de separação da parede celular da raiz no colmo da cana, será possível não só diminuir a quantidade de coquetéis enzimáticos usados hoje para fazer a hidrólise enzimática [degradação e conversão de carboidratos da palha e do bagaço da cana em açúcares capazes de sofrer fermentação] para obter o etanol de segunda geração como também aumentar a extração de sacarose”, disse Buckeridge, que também coordena o INCT do Bioetanol, à Agência FAPESP.
Foram sequenciados dois genes considerados essenciais nas etapas iniciais de desenvolvimento do aerênquima na raiz da cana-de-açúcar. O primeiro é o que codifica a proteína RAV (scRAV1), fator de transcrição que controla o envelhecimento das folhas das plantas. O segundo gene é o da endopoligalacturonase (scEPG1), enzima que ataca polissacarídeos (pectinas) que mantêm as células unidas e, dessa forma, realiza a separação celular durante o amadurecimento de frutos e a formação de aerênquima.
Devido à alta complexidade do genoma da cana, que possui várias cópias de cada cromossomo e numerosas variantes de cada gene, os dois genes foram sequenciados a partir de 17 clones cromossômicos bacterianos com regiões genômicas correspondentes a de uma variedade de cana, a R570.
As sequências dos genes foram comparadas com as do sorgo (Sorghum bicolor), uma vez que essa planta tem um dos genomas mais parecidos com o da cana. Uma sequência semelhante de cada um dos dois genes foi expressa em folhas de tabaco para ensaios de transativação – em que se avalia quais outros genes eles ativam.
“O tabaco é uma planta fácil de transformar geneticamente e serve como modelo para demonstrar se mecanismos como esses de fato funcionam”, disse Buckeridge.
As análises das regiões genômicas e dos ensaios de transativação demonstraram que o scRAV1 controla a degradação precoce de pectinas durante a formação do aerênquima da raiz da cana. Além disso, que a proteína produzida pelo gene scRAV1 se liga ao promotor do gene scEPG1, reprimindo sua transcrição.
“Isso abre a perspectiva de usar biotecnologia para fazer manipulação genética da cana e, com isso, poder aumentar a produção de etanol de segunda geração”, disse Buckeridge.
Primeiro passo
Os pesquisadores desenvolveram uma variedade de cana com expressão aumentada de scRAV1. Descobriram também um microRNA – molécula reguladora de expressão gênica – capaz de inibir especificamente o scRAV1 e estimular a expressão do scEPG1 em toda a planta.
“Estamos começando a ver nessa variedade transformada geneticamente o que parece ser um efeito de amolecimento da parede celular da cana próximo ao que queremos. O colmo de algumas delas, por exemplo, não consegue ficar ereto. Isso pode representar o primeiro passo para chegarmos ao desenvolvimento da cana papaia”, disse Buckeridge.
Um vídeo da tomografia da formação do aerênquima na raiz de cana pode ser visto em www.botany.one/2017/11/creative-destruction-death-leads-function.
O artigo The control of endopolygalacturonase expression by the sugarcane RAV transcription factor during aerenchyma formation (DOI: 10.1093/jxb/ery362), de Eveline Q. P. Tavares, Amanda P. de Souza, Grayce H. Romim, Adriana Grandis, Anna Plasencia, Jonas W. Gaiarsa, Jacqueline Grima-Pettenat, Nathalia de Setta, Marie-Anne Van Sluys e Marcos S. Buckeridge, pode ser lido no Journal of Experimental Botany em academic.oup.com/jxb/article/70/2/497/5272591?searchresult=1.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.





