
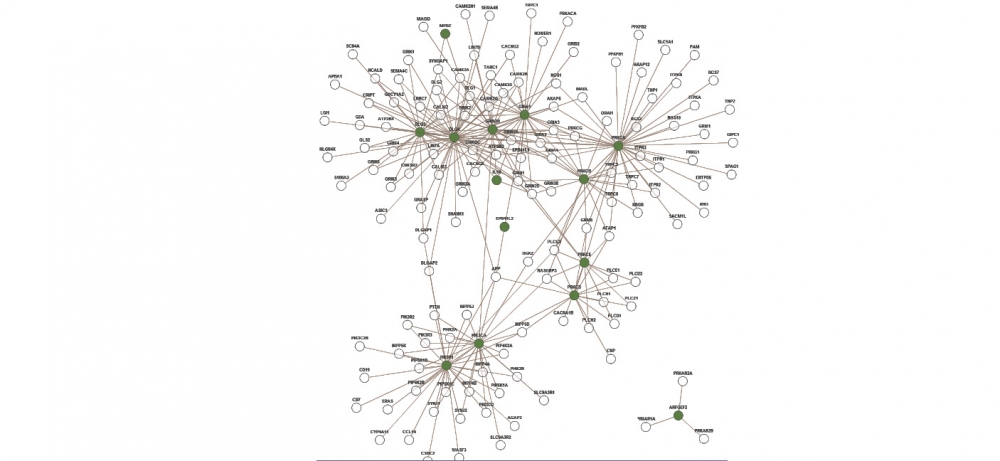
Rede da via glutamatérgica: genes responsáveis pela produção de 14 proteínas presentes na rede (nós em verde) foram identificados pela ferramenta como associados a esquizofrenia
Conceitos da teoria de grafos e de redes complexas, aliados a ferramentas como inferência de redes gênicas, ajudam a mapear aspectos estruturais e funcionais entre os genes e apontando alvos terapêuticos para doenças
Conceitos da teoria de grafos e de redes complexas, aliados a ferramentas como inferência de redes gênicas, ajudam a mapear aspectos estruturais e funcionais entre os genes e apontando alvos terapêuticos para doenças

Rede da via glutamatérgica: genes responsáveis pela produção de 14 proteínas presentes na rede (nós em verde) foram identificados pela ferramenta como associados a esquizofrenia
Karina Toledo, de Columbus (EUA) | Agência FAPESP – Ferramentas de bioinformática têm sido usadas por pesquisadores da Universidade Federal do ABC (UFABC) e da Universidade de São Paulo (USP) para modelar redes de interação entre genes, desvendar relações funcionais e, dessa forma, identificar potenciais alvos para o tratamento de doenças complexas, como esquizofrenia, autismo e câncer.
Avanços nessa área foram apresentados pelo professor do Centro de Matemática, Computação e Cognição (CMCC) da UFABC David Corrêa Martins Junior, no dia 31 de março, em Columbus, Estados Unidos, durante a programação da FAPESP Week Michigan-Ohio. O evento, que terminou na sexta-feira (1º/4), teve o objetivo de fomentar a colaboração entre pesquisadores paulistas e norte-americanos.
Parte dos resultados também foi publicada em artigos nos periódicos Information Sciences e BMC Bioinformatics.
"Uma mesma doença complexa pode se manifestar de formas muito diferentes em cada paciente, com diversos graus de gravidade. O objetivo desse tipo de estudo é poder oferecer um tratamento individualizado, considerando essas particularidades", explicou Martins Junior em entrevista à Agência FAPESP.
Os estudos são baseados tanto em dados clínicos – de expressão gênica, interação entre proteínas e metilação de DNA – depositados em bancos públicos, como em dados de pacientes atendidos por colaboradores, como, por exemplo, a professora Helena Brentani, do Instituto de Psiquiatria da USP, que trabalha com portadores de autismo.
Conceitos da teoria de grafos e de redes complexas, aliados a ferramentas de bioinformática, permitem, por exemplo, comparar em portadores de uma determinada doença e em indivíduos controle a expressão dos genes e sua centralidade nas redes gênicas. Também é possível comparar dados de pacientes com a mesma doença, mas com diferentes graus de gravidade, ou, ainda, avaliar como a expressão dos genes e a estrutura de uma rede gênica em uma mesma pessoa se modifica em diferentes contextos e momentos. A ideia é tentar desvendar como os genes estão conectados e como esse circuito controla as diversas funções celulares.
"Quando não sabemos a função de um gene, podemos tentar comparar a expressão dele ou a estrutura de sua vizinhança na rede gênica com outros que apresentam um sinal parecido e cuja função é conhecida e, assim, formular hipóteses. Se temos um gene-alvo e queremos saber em que medida a sua expressão depende do sinal de outros genes, podemos usar ferramentas como inferência de redes gênicas ou priorização gênica", contou Martins Junior
Segundo o pesquisador da UFABC, genes que já foram associados a doenças em estudos anteriores podem servir como ponto de partida para as análises. "Podemos avaliar quais outros genes estão mais associados a eles em termos de expressão e em termos da estrutura conectiva. Provavelmente eles também estarão envolvidos na doença em questão", disse.
Em um trabalho desenvolvido durante o doutorado de Sergio Nery Simões no programa de pós-graduação em Bioinformática da USP, foi desenvolvida uma ferramenta chamada NERI (NEtwork Medicine Relative Importance), que integra dados de expressão gênica, redes de interação entre proteínas e dados de estudos de associação (genes sabidamente associados a uma determinada doença) para identificar novos genes que também podem estar associados à enfermidade em análise.
"Fizemos um estudo de caso usando bancos de dados públicos de esquizofrenia e vimos que muitos dos genes obtidos pela ferramenta NERI estavam associados à chamada via glutamatérgica, responsável pela transmissão e recepção de neurotransmissores durante as sinapses neuronais. Tal via é bastante citada na literatura como associada à doença. Isso sugere que outros genes identificados pela ferramenta também têm potencial para estar relacionados com a esquizofrenia", explicou Martins Junior.
As pesquisas do grupo são apoiadas pela FAPESP por meio de dois Projetos Temáticos – um coordenado por João Eduardo Ferreira no Instituto de Matemática e Estatística (IME) da USP e outro por Roberto Marcondes Cesar Junior, também no IME-USP. Há ainda um Auxílio à Pesquisa – Regular coordenado por Raphael Yokoingawa de Camargo no CMCC-UFABC.
Análise de redes sociais
No mesmo painel dedicado ao tema "Bioinformática e Análise de Dados", o professor de Ciência da Computação e Engenharia da The Ohio State University Srinivasan Parthasarathy falou sobre como informações coletadas em redes sociais, como o Twitter ou o WhatsApp, podem ser usadas para ajudar a prever ou mitigar problemas relacionados a desastres naturais e epidemias.
Em parceria com pesquisadores de diversos países, inclusive brasileiros vinculados à Universidade Federal de Minas Gerais (UFMG), Parthasarathy coleta e analisa informações de mensagens curtas enviadas durante eventos climáticos extremos, como furacões e enchentes, ou durante surtos epidêmicos e usa o conteúdo para alimentar modelos computacionais. O objetivo é identificar problemas antes que eles se tornem críticos e fornecer informações que possam auxiliar as equipes que prestam socorro no local a planejar estratégias de controle.
"Se eu tenho 5 mil pessoas disponíveis para prestar socorro, preciso identificar as áreas mais afetadas e que precisam ser priorizadas. Para isso, usamos essas informações coletadas nas mídias sociais em conjunto com dados físicos que tradicionalmente alimentam os modelos de prevenção de furacão ou de enchentes, como velocidade do vento e pressão atmosférica, que são uma forma mais tradicional de sensoriamento", contou o pesquisador.
O grande desafio, segundo Parthasarathy, é conseguir filtrar as mensagens que realmente são relacionadas com o problema em questão. "Há muito ruído nas redes sociais. Por exemplo, se nosso objetivo é monitorar uma epidemia de dengue em uma determinada cidade, temos de ser capazes de isolar os tuítes que foram originados naquela região e incluir aquela informação em nosso modelo. Essa integração de sensores sociais e físicos pode produzir melhores resultados", comentou o pesquisador.
Também participaram do painel realizado durante a FAPESP Week Álvaro Montenegro, professor do Departamento de Geografia da The Ohio State University, e João Luiz Filgueiras de Azevedo, do Instituto de Aeronáutica e Espaço (IAE), do Departamento de Ciência e Tecnologia Aeroespacial (DCTA).
Para mais informações sobre a FAPESP Week Michigan-Ohio acesse http://www.fapesp.br/week2016/michigan-ohio/category/news/.
Republicar
A Agência FAPESP licencia notícias via Creative Commons (CC-BY-NC-ND) para que possam ser republicadas gratuitamente e de forma simples por outros veículos digitais ou impressos. A Agência FAPESP deve ser creditada como a fonte do conteúdo que está sendo republicado e o nome do repórter (quando houver) deve ser atribuído. O uso do botão HMTL abaixo permite o atendimento a essas normas, detalhadas na Política de Republicação Digital FAPESP.




