
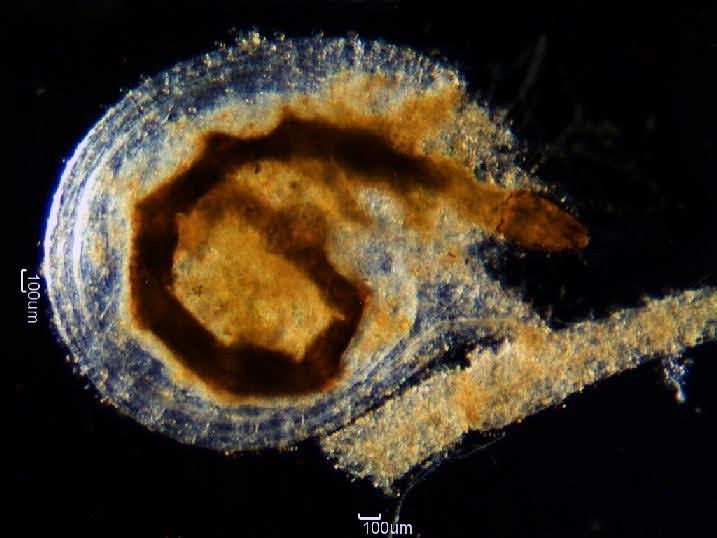
Científicos completan la primera secuenciación del ADNmt de una de las más de 120 especies de este grupo existentes en Brasil (foto: utrículo de Utricularia reniformis A.St.-Hil. con larva de artrópodo capturada/ divulgación)
Científicos completan la primera secuenciación del ADNmt de una de las más de 120 especies de este grupo existentes en Brasil
Científicos completan la primera secuenciación del ADNmt de una de las más de 120 especies de este grupo existentes en Brasil

Científicos completan la primera secuenciación del ADNmt de una de las más de 120 especies de este grupo existentes en Brasil (foto: utrículo de Utricularia reniformis A.St.-Hil. con larva de artrópodo capturada/ divulgación)
Por Peter Moon | Agência FAPESP – Existen aproximadamente 130 especies de plantas carnívoras en Brasil, que están presentes prácticamente en todos los biomas. Para comprender cómo se desarrolló la dinámica evolutiva de este grupo tan fascinante de plantas, una condición necesaria es el estudio de su ADN. Sin embargo, de ninguna de esas especies brasileñas se había secuenciado su genoma completo.
Y esto está empezando a cambiar a partir del trabajo de un grupo de la Universidade Estadual Paulista (Unesp), en Brasil, que está elaborando la primera secuenciación genómica total (cloroplasto, mitocondria y núcleo) de una planta carnívora brasileña, la Utricularia reniformis, endémica del país.
Los primeros resultados, con la publicación del plastoma –el genoma presente en los orgánulos denominados cloroplastos (cADN)– salieron publicados en 2016. Un segundo estudio, con el genoma mitocondrial (ADNmt), salió este año en la misma revista PLOS ONE. El próximo paso consistirá en la publicación del genoma nuclear (ADNn), que se encuentra en marcha.
Esta investigación está encabezada por el bioinformático Alessandro de Mello Varani y por el biólogo Vitor Fernandes Oliveira Miranda, ambos de la Facultad de Ciencias Agrarias y Veterinarias de la Unesp de Jaboticabal, en el estado de São Paulo, y cuenta con el apoyo de la FAPESP.
Las más de 120 especies de plantas carnívoras identificadas hasta el momento en Brasil no descienden de un ancestro común. Estas plantas evolucionaron en forma independiente en alrededor de nueve ocasiones distintas, en cinco órdenes diferentes de angiospermas.
El orden con la mayor cantidad de plantas carnívoras es Lamiales, el mismo de plantas tales como la lavanda, las lilas, los jazmines y la boca de dragón, aparte de otras especies de uso culinario como el olivo, el sésamo, la menta, la albahaca, la salvia y el romero.
Dentro del orden Lamiales, la familia con más especies carnívoras (370) es la de las lentibulariáceas, de las cuales alrededor de 230 especies pertenecen al género Utricularia, que congrega a plantas acuáticas y terrestres. La Utricularia reniformis es una planta terrestre que crece en lugares húmedos del Bosque Atlántico.
El método que utiliza la U. reniformis para capturar a sus presas consiste en una pequeña bolsa llamada utrículo. En su interior hay un fluido que sirve para digerir los microcrustáceos que eventualmente pueda succionar. Una vez apresados, no tienen escapatoria. La presa muere y sus tejidos son digeridos y absorbidos por la planta carnívora.
Pero todo esto sucede en una dimensión invisible para el ojo humano. “Los utrículos son sumamente pequeños y sólo es posible analizarlos en detalle en el microscopio”, dijo De Mello Varani.
“Se estima que los menores genomas de plantas son los de algunas especies de lentibulariáceas. Los genomas nucleares (ADNn) de los animales están más conservados, es decir, varían muy poco entre especies de un mismo género o de una misma familia en comparación con los genomas de las plantas”, dijo.
Los humanos y los chimpancés, por ejemplo, tienen una diferencia de tan sólo un 1,5% del genoma nuclear. Pero en el caso del ADN nuclear de las plantas de una misma especie provenientes de distintas poblaciones, la diferencia puede ser muy grande.
“Varían sobremanera dentro de un mismo género, e incluso entre distintos ejemplares de una misma especie. Entre las lentibulariáceas, por ejemplo, el tamaño del ADN nuclear puede variar de 61 millones a 1.600 millones de bases (nucleótidos) dentro de la misma familia. Hay especies de lentibulariáceas con genomas 25 veces menores que otras. Por lo tanto, las lentibulariáceas son excelentes candidatas para los estudios de contracción y expansión genómica”, dijo De Mello Varani.
Aparte de que el genoma de las plantas de un mismo género o de una misma especie varía en lo que hace a su tamaño, el orden en que están dispuestos los genes en las cadenas helicoidales de ADN también cambia. Es como si se agarraran los millones de nucleótidos del ADN nuclear de una planta, se los pusiera en una licuadora, se los mezclase en una nueva secuencia y el ADN resultante permaneciera funcional y de la misma especie.
“Los genomas de las plantas fluctúan en tamaño y en el orden de disposición de las bases en los cromosomas. Debido a esto, el trabajo con el genoma nuclear de plantas, desde el punto de vista bioinformático, es una tarea que constituye un desafío”, dijo De Mello Varani.
Es algo complicado, pero es posible, merced al avance de las técnicas de secuenciación y, fundamentalmente, de la bioinformática, la herramienta computacional utilizada para clasificar y comparar las decenas de bases de cada genoma. El desafío consiste en determinar que fragmentos del genoma corresponden a los genes funcionales.
“Es necesario determinar dónde empieza la secuencia de bases de un determinado gen y dónde termina, y descubrir, entre cadenas de ADN distintas extraídas de miembros de una misma especie, cuáles son los genes inherentes a dicha especie. En este caso específico, es necesario descubrir qué genes son los que determinan a la planta carnívora U. reniformis", dijo De Mello Varani.
El ADN del cloroplasto
Un factor que diferencia en gran medida la carga hereditaria completa de las plantas consiste en que, además del ADN nuclear y del ADN mitocondrial (ADNmt), aquél que está dentro de las mitocondrias (en los animales y en las plantas), estas últimas cuentan con un tercer reservorio genético que no existe en los animales. Se trata del plastoma, el ADN del cloroplasto (ADNcp), el orgánulo responsable de la fotosíntesis.
La teoría simbiótica indica que orgánulos tales como la mitocondria y el cloroplasto constituyen una herencia de antiguos microorganismos que hace miles de millones de años, en la era Proterozoica, invadieran o fueron absorbidos por bacterias mayores. En lugar de que uno destruyera a la otra o viceversa, se estableció entonces una relación simbiótica donde los ancestros bacterianos de las mitocondrias y los de los cloroplastos pasaron a desempeñar en el metabolismo del huésped el rol fundamental de la generación de energía: de los alimentos, en el caso de la mitocondria, y energía solar en el caso de los cloroplastos.
Como en un pasado remoto las mitocondrias y los cloroplastos fueron seres microscópicos independientes, estos orgánulos preservan en su interior secuencias de ADN que descienden directamente del ADN de aquellos pequeños invasores proterozoicos.
Cuando se habla de obtener el genoma completo de una planta, lo que se pretende es secuenciar tres ADNs: el ADN nuclear (ADNn) con decenas de millones de bases, el ADN mitocondrial (ADNmt) y el ADN del cloroplasto (ADNcp), con tan sólo algunos miles de bases cada uno.
“En términos de dificultades, la secuenciación del ADN del cloroplasto fue más fácil. Descubrimos muchos puntos interesantes, como el hecho de que muchos de los genes del cloroplasto de U. reniformis, que regulan la fotosíntesis, habían sido borrados o apagados del ADNcp. Fue un hallazgo interesante, toda vez que esos mismos genes se encuentran intactos en especies de hábitat acuático”, dijo De Mello Varani.
“Las plantas carnívoras hacen fotosíntesis. Pero esta condición carnívora es también una forma de obtener nutrientes. La pérdida de aquellos genes quizá tenga que ver con la adaptación a la misma y al hábitat terrestre de la planta; pero eso es algo que aún no lo sabemos”, dijo. La novedad que surgió de la secuenciación del ADN mitocondrial residió en descubrir que algunos de aquellos genes apagados en el cloroplasto fueron transportados y reconectados en la mitocondria. Pero esos genes aparentemente no se encuentran funcionales en el ADNmt.
“Ahora estamos estudiando la transferencia génica entre estos orgánulos y procurando entender ese proceso desde el punto de vista evolutivo. Comparamos el ADNcp y el ADNmt de U. reniformis con los genomas de las orgánulos de otras especies de hábitat terrestre del género y constatamos una tendencia a la pérdida de los genes del ADNcp y a la transferencia de los mismos al ADNmt en todas ellas. Lo interesante es que esta tendencia aparentemente no existe en especies de hábitat acuático, lo cual sugiere que las lentibulariáceas también pueden utilizarse como modelo para el estudio de la adaptación de las plantas a hábitats acuáticos”, dijo De Mello Varani.
Puede leerse el artículo intitulado The mitochondrial genome of the terrestrial carnivorous plant Utricularia reniformis (Lentibulariaceae): Structure, comparative analysis and evolutionary landmarks, de Saura R. Silva, Danillo O. Alvarenga, Yani Aranguren, Helen A. Penha, Camila C. Fernandes, Daniel G. Pinheiro, Marcos T. Oliveira, Todd P. Michael, Vitor F. O. Miranda y Alessandro M. Varani, en el siguiente enlace: doi.org/10.1371/journal.pone.0180484.
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.




