
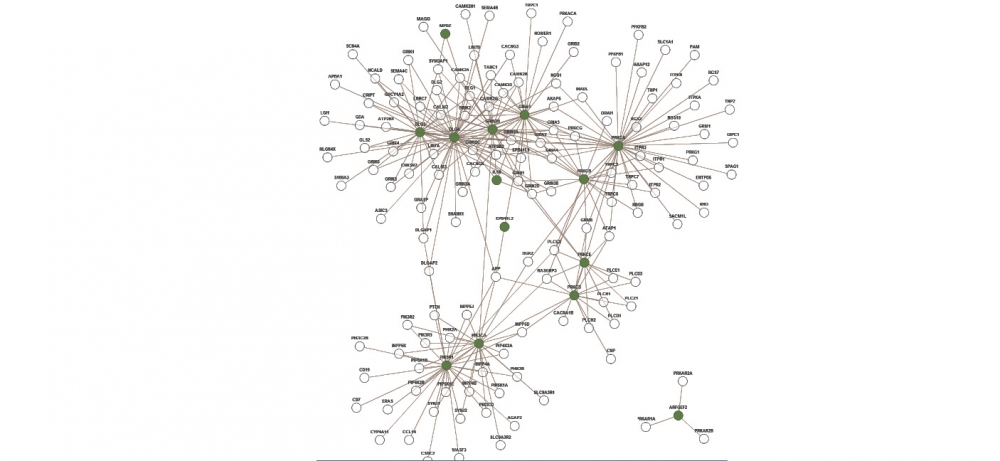
Un módulo de interacción entre proteínas existentes en humanos, responsables de la vía de señalización de receptores glutamatérgicos. Los genes encargados de la producción de 14 proteínas presentes en la red (los nodos en verde) se caracterizaron con esta herramienta como asociados a la esquizofrenia
Aliados a otras herramientas, a ejemplo de la inferencia de redes génicas, los conceptos de la teoría de grafos y redes complejas ayudan a mapear aspectos estructurales y funcionales de los genes
Aliados a otras herramientas, a ejemplo de la inferencia de redes génicas, los conceptos de la teoría de grafos y redes complejas ayudan a mapear aspectos estructurales y funcionales de los genes

Un módulo de interacción entre proteínas existentes en humanos, responsables de la vía de señalización de receptores glutamatérgicos. Los genes encargados de la producción de 14 proteínas presentes en la red (los nodos en verde) se caracterizaron con esta herramienta como asociados a la esquizofrenia
Por Karina Toledo, desde Ohio | Agência FAPESP – Las herramientas de la bioinformática ha sido utilizadas por científicos de la Universidad Federal del ABC (UFABC) y de la Universidad de São Paulo (USP) para modelar redes de interacción entre genes, develar relaciones funcionales y detectar así potenciales blancos para el tratamiento de enfermedades complejas, tales como la esquizofrenia, el autismo y el cáncer.
David Corrêa Martins Junior, docente del Centro de Matemática, Computación y Cognición (CMCC) de la UFABC, presentó avances en el área en Columbus, Estados Unidos, el día 31 de marzo, durante la programación de la FAPESP Week Michigan-Ohio. El evento, que culminó el viernes pasado (1° de abril), se realizó con el objetivo de fomentar la colaboración entre científicos paulistas y estadounidenses.
Parte de los resultados se publicó en artículos de las revistas científicas Information Sciences y BMC Bioinformatics.
“Una misma enfermedad compleja puede manifestarse de maneras muy distintas en cada paciente, con diversos grados de gravedad. El objetivo de este tipo de estudios consiste en poder ofrecer un tratamiento individualizado, que tenga en cuenta esas particularidades”, explicó Martins Junior en entrevista concedida a Agência FAPESP.
Los estudios se basan tanto en datos clínicos –de expresión génica, interacción entre proteínas y metilación de ADN– depositados en bancos públicos, como así también en datos de pacientes atendidos por colaboradores como la profesora Helena Brentani, del Instituto de Psiquiatría de la USP, por ejemplo, quien trabaja con portadores de autismo.
Conceptos de la teoría de grafos y de redes complejas, aliados a herramientas de bioinformática, permiten comparar la expresión de los genes y su centralidad en las redes génicas en portadores de una determinada enfermedad y en individuos de control, por ejemplo. También es posible comparar datos de pacientes con la misma enfermedad, pero en distintos grados de gravedad, o incluso evaluar de qué manera se modifica la expresión de los genes y la estructura de una red génica en una misma persona en diferentes contextos y momentos. La idea es intentar develar cómo se conectan los genes y cómo controlan ese circuito las diversas funciones celulares.
“Cuando no sabemos cuál es la función de un gen, podemos intentar comparar su expresión o la estructura aledaña al mismo en la red génica con otros que exhiben una señal parecida y cuya función se conoce, para así formular hipótesis. Si tenemos un gen blanco y pretendemos saber en qué medida su expresión depende de la señal de otros genes, podemos emplear herramientas tales como la inferencia de redes génicas o la priorización génica”, comentó Martins Junior.
Según el investigador de la UFABC, genes que ya fueron asociados a enfermedades en estudios anteriores pueden servir de punto de partida para la realización de nuevos análisis. “Podemos evaluar cuáles otros genes están más asociados a ellos en términos de expresión y de estructura conectiva. Probablemente éstos también estén involucrados en la enfermedad en cuestión”, dijo.
En un trabajo desarrollado durante el doctorado de Sergio Nery Simões, en el programa de posgrado en Bioinformática de la USP, se elaboró una herramienta llamada NERI (Network Medicine Relative Importance), que integra datos de expresión génica, redes de interacción entre proteínas y datos de estudios de asociación (genes sabidamente asociados con una determinada enfermedad) para detectar nuevos genes que también pueden estar asociados con la afección en análisis.
“Realizamos un estudio de caso utilizando bancos de datos públicos de esquizofrenia y observamos que muchos de los genes obtenidos con la herramienta NERI estaban asociados con la así llamada vía glutamatérgica, encargada de la transmisión y la recepción de neurotransmisores durante las sinapsis neuronales. Dicha vía es bastante citada en la literatura como asociada a la enfermedad. Esto sugiere que otros genes identificados con la herramienta también pueden potencialmente tener relación con la esquizofrenia”, explicó Martins Junior.
Las investigaciones del grupo cuentan con el apoyo de la FAPESP en el marco de dos Proyectos Temáticos, uno coordinado por João Eduardo Ferreira en el Instituto de Matemática y Estadística (IME) de la USP, y el otro por Roberto Marcondes Cesar Junior, también en el IME-USP. Y hay también una Ayuda a la Investigación – Regular, coordinada por Raphael Yokoingawa de Camargo en el CMCC-UFABC.
Análisis de redes sociales
En el mismo panel dedicado al tema “Bioinformática y Análisis de Datos”, el profesor de Ciencia de la Computación e Ingeniería de la The Ohio State University, Srinivasan Parthasarathy, se refirió a cómo puede utilizarse la información recabada en redes sociales tales como Twitter WhatsApp para ayudar a prever o mitigar problemas relacionados con desastres naturales y epidemias.
En colaboración con investigadores de diversos países, entre los cuales se incluyen brasileños vinculados a la Universidad Federal de Minas Gerais (UFMG), Parthasarathy recaba y analiza información referente a mensajes cortos enviadas durante eventos climáticos extremos, tales como huracanes e inundaciones, o durante brotes epidémicos, y utiliza su contenido para alimentar modelos computacionales. El objetivo consiste en detectar los problemas antes de que se vuelvan críticos y suministrar información que pueda ayudar a los equipos de auxilio locales en la planificación de estrategias de control.
“Si cuento con cinco mil personas dispuestas para a brindar auxilio, debo identificar cuáles son las áreas más afectadas para que constituyan la prioridad. A tal efecto, nos valemos de la información recabada en los medios sociales, junto con datos físicos que tradicionalmente se emplean para alimentar los modelos de prevención de huracanes o de inundaciones, tales como la velocidad del viento y la presión atmosférica, que constituyen una forma más tradicional de monitoreo”, comentó el investigador.
El gran reto, según Parthasarathy, consiste en conseguir filtrar los mensajes que efectivamente se relacionan con el problema en cuestión. “Hay mucho ruido en las redes sociales. Por ejemplo: si nuestro objetivo es monitorear una epidemia de dengue en una determinada ciudad, debemos ser capaces de aislar los tuits que se originaron en esa zona e incluir aquella información en nuestro modelo. Esta integración de sensores sociales y físicos puede producir mejores resultados”, comentó el investigador.
También participaron en el panel realizado durante la FAPESP Week Álvaro Montenegro, docente del Departamento de Geografía de la The Ohio State University, y João Luiz Filgueiras de Azevedo, del Instituto de Aeronáutica y Espacio (IAE).
Republicar
The Agency FAPESP licenses news via Creative Commons (CC-BY-NC-ND) so that they can be republished free of charge and in a simple way by other digital or printed vehicles. Agência FAPESP must be credited as the source of the content being republished and the name of the reporter (if any) must be attributed. Using the HMTL button below allows compliance with these rules, detailed in Digital Republishing Policy FAPESP.




